|
Kilka tysięcy praojców
R1a1a1-M417* i H14a |
Skąd
pochodzą Dyskusja o mutacjach STR. Tablice |
Ostatnia
aktualizacja
|
†
WYBIERZ RÓD:
|
nadto: GIERAŁTOWIE |
GENEALOGIA GENETYCZNA. |
Strony genealogii genetycznej na naszej witrynie
1.
Genealogia Y i mtDNA, cz.1, haplogrupy R1a, R1b
2.
Geneza języków
indoeuropejskich i początek Słowian
3.
Haplogrupy nieindoeuropejskie
Y-DNA. Haplogrupy mitochondrialne mtDNA
Jak zbadać swoje Y-DNA i
mtDNA
Pochodzenie ludności w dorzeczu Dunajca i jego sąsiedztwie * Inne wybrane regiony
Ilustracje R1a1a: drzewa, mapy, filogeneza
Skąd, Europejczyku, jak i kiedy tu przybyłeś?
Nowiny i linki do naukowych publikacji
Rozmaitości genealogii genetycznej i dyskusja [cz.1] [cz.2]
Dyskusja o mutacjach STR. Faktyczny czas mutacji. TABLICE
Y-Adam, praojciec wszystkich współczesnych ludzi [1]
[2] *
Fundusz RRA00
[1]
[2]
Kreacjonizm
ewolucjonistyczny
The evolutionistic
creationism
Uwaga. Stan dyskusji w latach 2010-2012, więc
nieaktualny !!! (StP)
1. Mutacje STR i ich tempo w markerach
W genealogii Y-DNA brane są pod uwagę mutacje w kilkudziesięciu szybkozmiennych miejscach DNA, zwanych markerami. Są to zmiany, polegające na wypadnięciu, dodaniu lub wymianie jednego z elementów powtarzającego się krótkiego tandemu (układu, sekwencji) zasad: A (adeniny), C (cytozyny), G (guaniny) i T (tiaminy). Na skutek takiej zmiany seria powtórzeń krótkiego tandemu (Short Tandem Repeats, STR) skraca się albo wydłuża. Liczba powtórzeń w danym miejscu (markerze) nazywana jest allelem.
Zestaw kilkunastu lub kilkudziesięciu, np. 12, 24, 37 lub 67 alleli stanowi haplotyp Y-DNA danej osoby. O ile nie ma laboratoryjnie rozpoznanej mutacji SNP, wtedy właśnie liczba i rodzaj zaistniałych i przekazanych potomkom zmian, czyli mutacji w haplotypie, mogą stanowić narzędzie rozpoznania haplogrupy, czyli rodu genetycznego i obliczania czasu, jaki upłynął od wspólnego przodka kilku lub większej grupy osób. Dlatego ważna jest znajomość częstotliwości, czyli czasu powstawania owych mutacji.
1a. Tabela tempa mutacji w 67 markerach STR (wg FTDNA)
http://freepages.genealogy.rootsweb.ancestry.com/~geneticgenealogy/ratestuff.htm
(z przeliczeniem liczby mutacji w czasie 307125 lat w
najbardziej powolnym DYS578 - stp.)
| Years per Mutation @ 25 yrs/gen |
% of descendant haplotypes with at least 1 mutation after x years |
|||||||||||||||
|
Note: The 95% CI's for the mutation rates are appx -50% and +100%. This affects the following %'s accordingly. |
||||||||||||||||
| # | locus |
rate |
gen/mut |
yr/mut |
500 | 1000 | 2000 | 3000 | 4000 | 5000 | 6000 | 7000 | 8000 | 9000 | 10000 |
na lat |
| 1 | 393 | 0.00076 | 1316 | 32895 | 2% | 3% | 6% | 9% | 12% | 15% | 18% | 21% | 24% | 27% | 30% |
9,34 |
| 2 | 390 | 0.00311 | 322 | 8039 | 6% | 12% | 25% | 37% | 50% | 62% | 75% | 87% | 100% | 112% | 124% |
38,20 |
| 3 | 19 | 0.00151 | 662 | 16556 | 3% | 6% | 12% | 18% | 24% | 30% | 36% | 42% | 48% | 54% | 60% |
18,55 |
| 4 | 391 | 0.00265 | 377 | 9434 | 5% | 11% | 21% | 32% | 42% | 53% | 64% | 74% | 85% | 95% | 106% |
32,56 |
| 5 | 385a | 0.00226 | 442 | 11062 | 5% | 9% | 18% | 27% | 36% | 45% | 54% | 63% | 72% | 81% | 90% |
27,76 |
| 6 | 385b | 0.00226 | 442 | 11062 | 5% | 9% | 18% | 27% | 36% | 45% | 54% | 63% | 72% | 81% | 90% |
27,76 |
| 7 | 426 | 0.00009 | 11111 | 277778 | 0% | 0% | 1% | 1% | 1% | 2% | 2% | 3% | 3% | 3% | 4% |
1,11 |
| 8 | 388 | 0.00022 | 4545 | 113636 | 0% | 1% | 2% | 3% | 4% | 4% | 5% | 6% | 7% | 8% | 9% |
2,70 |
| 9 | 439 | 0.00477 | 210 | 5241 | 10% | 19% | 38% | 57% | 76% | 95% | 114% | 134% | 153% | 172% | 191% |
58,60 |
| 10 | 389i | 0.00186 | 538 | 13441 | 4% | 7% | 15% | 22% | 30% | 37% | 45% | 52% | 60% | 67% | 74% |
22,85 |
| 11 | 392 | 0.00052 | 1923 | 48077 | 1% | 2% | 4% | 6% | 8% | 10% | 12% | 15% | 17% | 19% | 21% |
6,39 |
| 12 | 389ii | 0.00242 | 413 | 10331 | 5% | 10% | 19% | 29% | 39% | 48% | 58% | 68% | 77% | 87% | 97% |
29,73 |
| 13 | 458 | 0.00814 | 123 | 3071 | 16% | 33% | 65% | 98% | 130% | 163% | 195% | 228% | 260% | 293% | 326% |
100,01 |
| 14 | 459a | 0.00132 | 758 | 18939 | 3% | 5% | 11% | 16% | 21% | 26% | 32% | 37% | 42% | 48% | 53% |
16,22 |
| 15 | 459b | 0.00132 | 758 | 18939 | 3% | 5% | 11% | 16% | 21% | 26% | 32% | 37% | 42% | 48% | 53% |
16,22 |
| 16 | 455 | 0.00016 | 6250 | 156250 | 0% | 1% | 1% | 2% | 3% | 3% | 4% | 4% | 5% | 6% | 6% |
1,97 |
| 17 | 454 | 0.00016 | 6250 | 156250 | 0% | 1% | 1% | 2% | 3% | 3% | 4% | 4% | 5% | 6% | 6% |
1,97 |
| 18 | 447 | 0.00264 | 379 | 9470 | 5% | 11% | 21% | 32% | 42% | 53% | 63% | 74% | 84% | 95% | 106% |
32,43 |
| 19 | 437 | 0.00099 | 1010 | 25253 | 2% | 4% | 8% | 12% | 16% | 20% | 24% | 28% | 32% | 36% | 40% |
12,16 |
| 20 | 448 | 0.00135 | 741 | 18519 | 3% | 5% | 11% | 16% | 22% | 27% | 32% | 38% | 43% | 49% | 54% |
16,58 |
| 21 | 449 | 0.00838 | 119 | 2983 | 17% | 34% | 67% | 101% | 134% | 168% | 201% | 235% | 268% | 302% | 335% |
102,96 |
| 22 | 464a | 0.00566 | 177 | 4417 | 11% | 23% | 45% | 68% | 91% | 113% | 136% | 158% | 181% | 204% | 226% |
69,53 |
| 23 | 464b | 0.00566 | 177 | 4417 | 11% | 23% | 45% | 68% | 91% | 113% | 136% | 158% | 181% | 204% | 226% |
69,53 |
| 24 | 464c | 0.00566 | 177 | 4417 | 11% | 23% | 45% | 68% | 91% | 113% | 136% | 158% | 181% | 204% | 226% |
69,53 |
| 25 | 464d | 0.00566 | 177 | 4417 | 11% | 23% | 45% | 68% | 91% | 113% | 136% | 158% | 181% | 204% | 226% |
69,53 |
| Years per Mutation @ 25 yrs/gen |
% of descendant haplotypes with at least 1 mutation after x years |
|||||||||||||||
| # | locus | rate | gen/mut | yr/mut | 500 | 1000 | 2000 | 3000 | 4000 | 5000 | 6000 | 7000 | 8000 | 9000 | 10000 | |
| 26 | 460 | 0.00402 | 249 | 6219 | 8% | 16% | 32% | 48% | 64% | 80% | 96% | 113% | 129% | 145% | 161% |
49,38 |
| 27 | H4 | 0.00208 | 481 | 12019 | 4% | 8% | 17% | 25% | 33% | 42% | 50% | 58% | 67% | 75% | 83% |
25,55 |
| 28 | YCAIIa | 0.00123 | 813 | 20325 | 2% | 5% | 10% | 15% | 20% | 25% | 30% | 34% | 39% | 44% | 49% |
15,11 |
| 29 | YCAIIb | 0.00123 | 813 | 20325 | 2% | 5% | 10% | 15% | 20% | 25% | 30% | 34% | 39% | 44% | 49% |
15,11 |
| 30 | 456 | 0.00735 | 136 | 3401 | 15% | 29% | 59% | 88% | 118% | 147% | 176% | 206% | 235% | 265% | 294% |
90,30 |
| 31 | 607 | 0.00411 | 243 | 6083 | 8% | 16% | 33% | 49% | 66% | 82% | 99% | 115% | 132% | 148% | 164% |
50,49 |
| 32 | 576 | 0.01022 | 98 | 2446 | 20% | 41% | 82% | 123% | 164% | 204% | 245% | 286% | 327% | 368% | 409% |
125,56 |
| 33 | 570 | 0.00790 | 127 | 3165 | 16% | 32% | 63% | 95% | 126% | 158% | 190% | 221% | 253% | 284% | 316% |
97,04 |
| 34 | CDYa | 0.03531 | 28 | 708 | 71% | 141% | 282% | 424% | 565% | 706% | 847% | 989% | 1130% | 1271% | 1412% |
433,79 |
| 35 | CDYb | 0.03531 | 28 | 708 | 71% | 141% | 282% | 424% | 565% | 706% | 847% | 989% | 1130% | 1271% | 1412% |
433,79 |
| 36 | 442 | 0.00324 | 309 | 7716 | 6% | 13% | 26% | 39% | 52% | 65% | 78% | 91% | 104% | 117% | 130% |
39,80 |
| 37 | 438 | 0.00055 | 1818 | 45455 | 1% | 2% | 4% | 7% | 9% | 11% | 13% | 15% | 18% | 20% | 22% |
6,76 |
| 38 | 531 | 0.00037 | 2669 | 66724 | 1% | 1% | 3% | 4% | 6% | 7% | 9% | 10% | 12% | 13% | 15% |
4,60 |
| 39 | 578 | 0.00008 | 12285 | 307125 | 0% | 0% | 1% | 1% | 1% | 2% | 2% | 2% | 3% | 3% | 3% |
1,00 |
| 40 | 395s1 | 0.00031 | 3239 | 80982 | 1% | 1% | 2% | 4% | 5% | 6% | 7% | 9% | 10% | 11% | 12% |
3,79 |
| 41 | 395s1 | 0.00031 | 3239 | 80982 | 1% | 1% | 2% | 4% | 5% | 6% | 7% | 9% | 10% | 11% | 12% |
3,79 |
| 42 | 590 | 0.00054 | 1837 | 45916 | 1% | 2% | 4% | 7% | 9% | 11% | 13% | 15% | 17% | 20% | 22% |
6,69 |
| 43 | 537 | 0.00057 | 1740 | 43508 | 1% | 2% | 5% | 7% | 9% | 11% | 14% | 16% | 18% | 21% | 23% |
7,06 |
| 44 | 641 | 0.00018 | 5647 | 141171 | 0% | 1% | 1% | 2% | 3% | 4% | 4% | 5% | 6% | 6% | 7% |
2,18 |
| 45 | 472 | 0.00001 | 67981 | 1699524 | 0% | 0% | 0% | 0% | 0% | 0% | 0% | 0% | 0% | 1% | 1% |
- |
| 46 | 406s1 | 0.00154 | 651 | 16282 | 3% | 6% | 12% | 18% | 25% | 31% | 37% | 43% | 49% | 55% | 61% |
18,86 |
| 47 | 511 | 0.00128 | 783 | 19581 | 3% | 5% | 10% | 15% | 20% | 26% | 31% | 36% | 41% | 46% | 51% |
15,68 |
| 48 | 425 | 0.00018 | 5623 | 140584 | 0% | 1% | 1% | 2% | 3% | 4% | 4% | 5% | 6% | 6% | 7% |
2,18 |
| 49 | 413 | 0.00202 | 494 | 12358 | 4% | 8% | 16% | 24% | 32% | 40% | 49% | 57% | 65% | 73% | 81% |
24,85 |
| 50 | 413 | 0.00202 | 494 | 12358 | 4% | 8% | 16% | 24% | 32% | 40% | 49% | 57% | 65% | 73% | 81% |
24,85 |
| Years per Mutation @ 25 yrs/gen |
% of descendant haplotypes with at least 1 mutation after x years |
|||||||||||||||
| # | locus | rate | gen/mut | yr/mut | 500 | 1000 | 2000 | 3000 | 4000 | 5000 | 6000 | 7000 | 8000 | 9000 | 10000 | |
| 51 | 557 | 0.00321 | 312 | 7796 | 6% | 13% | 26% | 38% | 51% | 64% | 77% | 90% | 103% | 115% | 128% |
39,39 |
| 52 | 594 | 0.00029 | 3504 | 87602 | 1% | 1% | 2% | 3% | 5% | 6% | 7% | 8% | 9% | 10% | 11% |
3,51 |
| 53 | 436 | 0.00018 | 5569 | 139237 | 0% | 1% | 1% | 2% | 3% | 4% | 4% | 5% | 6% | 6% | 7% |
2,21 |
| 54 | 490 | 0.00019 | 5322 | 133049 | 0% | 1% | 2% | 2% | 3% | 4% | 5% | 5% | 6% | 7% | 8% |
2,31 |
| 55 | 534 | 0.00832 | 120 | 3005 | 17% | 33% | 67% | 100% | 133% | 166% | 200% | 233% | 266% | 299% | 333% |
102,20 |
| 56 | 450 | 0.00020 | 4903 | 122579 | 0% | 1% | 2% | 2% | 3% | 4% | 5% | 6% | 7% | 7% | 8% |
2,51 |
| 57 | 444 | 0.00321 | 312 | 7793 | 6% | 13% | 26% | 38% | 51% | 64% | 77% | 90% | 103% | 115% | 128% |
39,41 |
| 58 | 481 | 0.00544 | 184 | 4596 | 11% | 22% | 44% | 65% | 87% | 109% | 131% | 152% | 174% | 196% | 218% |
66,82 |
| 59 | 520 | 0.00245 | 408 | 10188 | 5% | 10% | 20% | 29% | 39% | 49% | 59% | 69% | 79% | 88% | 98% |
30,15 |
| 60 | 446 | 0.00365 | 274 | 6844 | 7% | 15% | 29% | 44% | 58% | 73% | 88% | 102% | 117% | 132% | 146% |
44,88 |
| 61 | 617 | 0.00042 | 2363 | 59074 | 1% | 2% | 3% | 5% | 7% | 8% | 10% | 12% | 14% | 15% | 17% |
5,20 |
| 62 | 568 | 0.00053 | 1901 | 47514 | 1% | 2% | 4% | 6% | 8% | 11% | 13% | 15% | 17% | 19% | 21% |
6,46 |
| 63 | 487 | 0.00097 | 1032 | 25801 | 2% | 4% | 8% | 12% | 16% | 19% | 23% | 27% | 31% | 35% | 39% |
11,90 |
| 64 | 572 | 0.00212 | 472 | 11795 | 4% | 8% | 17% | 25% | 34% | 42% | 51% | 59% | 68% | 76% | 85% |
26,04 |
| 65 | 640 | 0.00034 | 2956 | 73901 | 1% | 1% | 3% | 4% | 5% | 7% | 8% | 9% | 11% | 12% | 14% |
4,16 |
| 66 | 492 | 0.00042 | 2363 | 59074 | 1% | 2% | 3% | 5% | 7% | 8% | 10% | 12% | 14% | 15% | 17% |
5,20 |
| 67 | 565 | 0.00087 | 1144 | 28610 | 2% | 3% | 7% | 10% | 14% | 17% | 21% | 24% | 28% | 31% | 35% |
10,73 |
| # | locus | rate | gen/mut | yr/mut | 500 | 1000 | 2000 | 3000 | 4000 | 5000 | 6000 | 7000 | 8000 | 9000 | 10000 | |
|
R A Z E M M U T A C J I W H A P L O T Y P I E 67 - M A R K E R O W Y M |
2755,48 |
|||||||||||||||
1b. Tabela tempa mutacji w markerach 68-111 (wg FTDNA)
Na podstawie: S.Paterson, maj 2011 ( http://dl.dropbox.com/u/2733445/MODEL1.xlsx )
Zob. J.Chandler, w: RootsWeb, 27.06.2011 (z przeliczeniem ilości mutacji; stp.)
|
Tempo mutacji w markerach od 68-111 |
|||
|
L o c u s |
Marker |
Liczba |
Ilość |
|
A |
B |
C |
D |
|
68 |
DYS710 |
0,01722 |
344,4 |
|
69 |
DYS485 |
0,00204 |
40,8 |
|
70 |
DYS632 |
0,00001 |
- |
|
71 |
DYS495 |
0,00135 |
27,0 |
|
72 |
DYS540 |
0,00077 |
15,4 |
|
73 |
DYS714 |
0,00502 |
100,4 |
|
74 |
DYS716 |
0,00042 |
8,4 |
|
75 |
DYS717 |
0,00265 |
53,0 |
|
76 |
DYS505 |
0,00355 |
71,0 |
|
77 |
DYS556 |
0,00142 |
28,4 |
|
78 |
DYS549 |
0,00432 |
86,4 |
|
79 |
DYS589 |
0,00192 |
48,4 |
|
80 |
DYS522 |
0,00103 |
20,6 |
|
81 |
DYS494 |
0,00008 |
1,6 |
|
82 |
DYS533 |
0,00362 |
72,4 |
|
83 |
DYS636 |
0,00099 |
19,8 |
|
84 |
DYS575 |
0,00018 |
3,6 |
|
85 |
DYS638 |
0,00063 |
12,6 |
|
86 |
DYS462 |
0,00046 |
9,2 |
|
87 |
DYS452 |
0,00223 |
44,6 |
|
88 |
DYS445 |
0,00082 |
16,4 |
|
89 |
Y-GATA-A10 |
0,00278 |
55,6 |
|
90 |
DYS463 |
0,00095 |
19,0 |
|
91 |
DYS441 |
0,00238 |
47,6 |
|
92 |
Y-ggaatt |
0,00081 |
16,2 |
|
93 |
DYS525 |
0,00121 |
24,2 |
|
94 |
DYS712 |
0,02194 |
438,8 |
|
95 |
DYS593 |
0,00022 |
4,4 |
|
96 |
DYS650 |
0,00662 |
132,4 |
|
97 |
DYS532 |
0,00259 |
51,8 |
|
98 |
DYS715 |
0,00364 |
72,8 |
|
99 |
DYS504 |
0,00414 |
82,8 |
|
100 |
DYS513 |
0,00275 |
55,0 |
|
101 |
DYS561 |
0,00198 |
39,6 |
|
102 |
DYS552 |
0,00253 |
50,6 |
|
103 |
DYS726 |
0,00010 |
2,0 |
|
104 |
DYS635 |
0,00373 |
74,6 |
|
105 |
DYS587 |
0,00097 |
19,4 |
|
106 |
DYS643 |
0,00180 |
36,0 |
|
107 |
DYS497 |
0,00093 |
18,6 |
|
108 |
DYS510 |
0,00298 |
59,6 |
|
109 |
DYS434 |
0.00018 |
3,6 |
|
110 |
DYS461 |
0,00210 |
42,0 |
|
111 |
DYS435 |
0,00005 |
1,0 |
|
Razem mutacji w markerach 68-111: |
2373,0 |
||
1c. Stosowane stopy mutacji w poszczególnych haplotypach
(zwłaszcza w środowisku Rosyjskiej Akademii Genealogii DNA)
|
Rodzaj |
Liczba mutacji |
Liczba mutacji |
Liczba lat |
|
12 markerów |
0,00183 |
0,022 |
1136 |
|
25 markerów |
0,00184 |
0,046 |
543 |
|
37 markerów |
0,00243 |
0,090 |
278 |
|
67 markerów |
0,00179 |
0,120 |
111 |
|
111 markerów |
0,00150 |
0,165 |
150 |
Stosowanie powyższych stóp mutacji wymaga uzupełnienia danymi z tablicy tzw.
mutacji zwrotnych (powtórnych w tym samym markerze),
których ilość zależy od liczby liczby tzw. mutacji prostych. Tablica ta jednak
uwzględnia nieznaczną część mutacji powtórnych i daje wyniki znacznie zaniżone
(zob. w Dyskusji). Stąd zachodzi potrzeba
innego systemu obliczania czasu mutacji.
2. Nowy sposób liczenia czasu mutacji STR
Założeniem poniższej tablicy jest fakt, że obserwowane
(widoczne) mutacje powstają w dowolnej kolejności markerów.
Statystycznie, jako widoczne ujawniają się zasadniczo tylko mutacje
pierwsze w danym markerze.
Równocześnie w markerach już zmutowanych dochodzi do licznych mutacji
powtórnych, stosownie do tempa mutacji w danym markerze. Mutacje nie są jednokierunkowe, lecz (co do liczby
alleli w markerze) mogą być na równi "w górę" lub "w dół", więc jakby drepczą w
miejscu; dlatego mutacje powtórne można statystycznie uważać za niewidoczne.
Czas mutacji widocznych w haplotypie nie dodaje się w sposób prosty (jak to praktykują niektórzy
genealodzy Y-DNA), czyli jeżeli pierwsza mutacja (stopa mutacji) powstanie np. w
czasie około 111 lat, to nie oznacza, że trzy mutacje pojawią się około 333
lata. O opóźnianiu się kolejnych mutacji decyduje ilość już zmutowanych i niezmutowanych markerów w haplotypie oraz równoczesne "namnażanie się"
mutacji powtórnych, statystycznie niewidocznych.
Dlatego Rosyjska Akademia DNA-Genealogii posługuje się
podwójną tablicą: jedna dla obliczania czasu mutacji prostych, a druga - mutacji
powtórnych ("zwrotnych"), ale niestety o stawkach zaniżonych. Inni ustalają
tzw. współczynnik ewolucyjny, jeden, niedopasowany ani do czasu bliskiego
(zawyżony), ani do czasów bardzo odległych (zaniżony).
3. Pełny czas mutacji STR.
Podręczna i praktyczna tabela PCM.
Full Times of Mutations STR.
The practical table FTM
( ulepszono / improved / 10.08.2012 - stp.)
|
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Uwaga. Obliczanie czasu wymaga umiejętności ustalania liczby mutacji
i genetycznego dystansu do najbliższego wspólnego przodka – MRCA
Attention. Time calculation requires counting skills mutations
and genetic distance to the recent common ancestor - MRCA)
Powyższą tabelę PCM dla 67 markerowego haplotypu można także
stosować
- dla mutacji w haplotypie 25 markerowym, mnożąc je przez
2,65
- dla mutacji w haplotypie 37 markerowym, mnożąc je przez
1,50
- dla mutacji w haplotypie 111 markerowym, dzieląc je przez
1,75
Uwaga
Środkowa kolumna (B) tej tabeli
służy tylko uzasadnieniu wyniku.
3a. Zasady powstania podręcznej i
praktycznej tabeli
pełnego czasu mutacji ( PCM )
Tabela PCM powstała na podstawie następujących czynników
1. Podstawą obliczeń w tabeli jest statystycznie domiemana obecność jednej mutacji w najbardziej wolno mutującym markerze, DYS425 = 500000 lat, a tym samym i w pozostałych markerach tego haplotypu.
(The basis of calculation in the table is statistically determined the presence of one mutation in the most slow mutating marker, DYS425 = 500,000 years, and thus, and other markers of this haplotype).
2. Ciąg wykładniczy (geometryczny) mutacji widocznych jest oparty na fakcie, że w haplotypie, wraz z kurczeniem się od 67/1 do 1/67 miejsc jeszcze niezmutowanych, odwrotnie wzrasta od 1/67 do 67/1 ilość miejsc na mutacje powtórne, czyli statystycznie niewidoczne (na miejsce mutacji widocznych powstają niewidoczne).
Według publikowanych zwłaszcza przez FTDNA (i J.Chandlera) tabel mutacji w parach ojciec-syn, do mutacji STR w najbardziej powolnym markerze DYS425 (z poprawką Klosowa) w 66 markerowym haplotypie (po zrezygnowaniu z jeszcze bardziej powolnego DYS472) dochodzi statystycznie co 500.000 lat. Tymczasem, biorąc pod uwagę obliczone z tabeli J. Chandlera tempo mutacji w haplotypie: jedna mutacja na około 111 lat, do statystycznego czasu zaistnienia tej mutacji DYS425, a tym samym i całego zestawu 66 mutacji widocznych, powstaje około 4505 mutacji.
Tak więc w czasie 66 mutacji widocznych, dochodzi także, w markerach już zmutowanych, do bardzo licznych mutacji powtórnych, czyli 4439 statystycznie niewidocznych, odpowiednio opóźniających czas owych mutacji widocznych, w sumie około 68,25 razy. Mutacje widoczne spowalniają się, gdyż w ich miejsce powstają wtedy mutacje powtóre, czyli statystycznie niewidoczne.
To spowolnianie tempa mutacji widocznych, spowodowane kurczeniem się dla nich miejsc dotąd niezmutowanych oraz nierównością tempa mutacji w poszczególnych markerach od jeden do około kilkadziesiąt lub kilkaset, trzeba dla cełów obliczeniowych wyrazić ciągiem wykładniczym. Dlatego, aby otrzymać pełny czas mutacji ( PCM ) dla każdej liczby mutacji w tym 67 minus 1 markerowym haplotypie, należy znaleźć iloraz ciągu wykładniczego, tj. “a” w funkcji wykładniczej a x (1...65). Tym ilorazem a jest uwidoczniona na tabeli PCM liczba 1,06713.
Uwaga
Powyższa zasada obliczania pełnego czasu mutacji (PCM),
uwzględniająca pełny zestaw mutacji widocznych i niewidocznych (powtórnych)
-
a) nie wymaga osobnych tablic na tzw. mutacje zwrotne (zob. w
tablicach A.Klosowa);
b) nie wymaga stosowania dla czasów "ewolucyjnych" (np. powyżej
10000 lat) dodatkowej tabeli 22-markerowego haplotypu z wybranych
markerów powolnych (zob. tablica Klosowa);
c) nie wymaga wreszcie tzw. ewolucyjnego "współczynnika
Żywotovskyego" (0,000069 mutacji na marker na pokolenie), który przy
zastosowaniu go do czasów bliskich, genealogicznych, zawyżając rezultat
niekiedy aż trzykrotnie, wywołuje wśród genealogów i genetyków tak wiele
sprzeciwu i krytyki.
Równocześnie tablica PCM stwarza możliwość obliczania czasu
najstarszych haplogrup i populacji, praktycznie od początku
współczesnego człowieka.
3b. Krzywa wykładnicza czasu mutacji widocznych
The exponential curve of time visible mutations
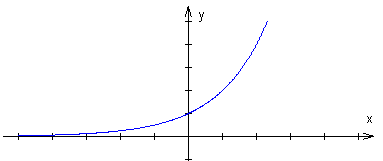
Wzrasta ogólna liczba mutacji i ich czas Y
ale zarazem spowolnia się i zatrzymuje przyrost mutacji
widocznych X
3c. Analogia - w krzywej wykładniczej czasu widocznych SNP
w/g modelu Jukesa-Cantora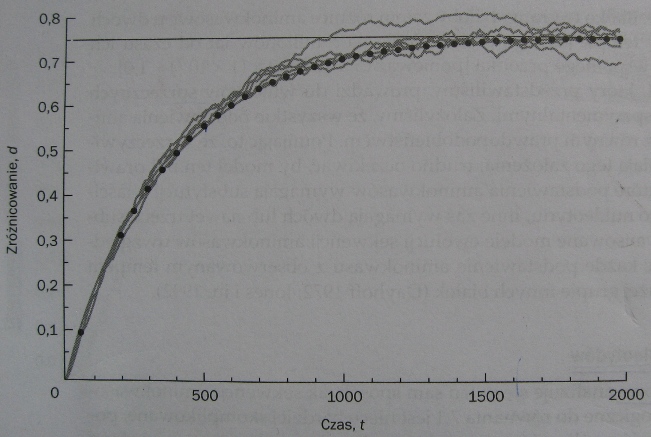
(Źródło wykresu: Podręcznik genetyki)
Model Jukesa-Cantora (1969). Model J-C ilustruje fakt, że wraz ze wzrostem czasu t i ogólnej liczby mutacji ogranicza się, a w końcu całkowicie zatrzymuje się wzrost mutacji widocznych na rzecz niewidocznych, czyli widoczne zróżnicowanie d w badanym odcinku Y-DNA (NRY). Dalszemu wzrostowi czasu nie odpowiada już żaden przyrost liczby mutacji widocznych.
Dlatego nie ma sensu stosowanie liniowego liczenia czasu
mutacji w haplotypie i stałego współczynnika czasu mutacji dla każdej
widocznej mutacji, co niektórzy genetycy niestety praktykują.
...............................................................................................
................................................................................
..............................................................................................
................................................................................
4. Tabela 21 powolnych markerów
w haplotypie 67 markerowym
Table 21 slow markers in 67-markers haplotype
(w 1. i 2. kolumnie wykorzystano markery i poprawki tempa ich mutacji od A. Klosowa)
|
Locus |
mut./25 lat |
mut./rok |
czas 1 mut. |
ilość mutacji |
|
DYS425 |
0,00005 |
0,0000020 |
500 000 |
1,00 |
|
DYS436 |
0,00006 |
0,0000024 |
416 667 |
1,12 |
|
DYS426 |
0,00009 |
0,0000036 |
277 778 |
1,80 |
|
DYS490 |
0,00013 |
0,0000050 |
200 000 |
2,50 |
|
DYS454 |
0,00016 |
0,0000064 |
156 250 |
3,20 |
|
DYS455 |
0,00016 |
0,0000064 |
156 250 |
3,20 |
|
DYS578 |
0,00017 |
0,0000068 |
147 059 |
3,40 |
|
DYS641 |
0,00017 |
0,0000068 |
147 059 |
3,40 |
|
DYS590 |
0,00017 |
0,0000068 |
147 059 |
3,40 |
|
DYS594 |
0,00020 |
0,0000080 |
125 000 |
4,00 |
|
DYS388 |
0,00022 |
0,0000088 |
113 636 |
4,40 |
|
DYS492 |
0,00023 |
0,0000092 |
108 696 |
4,60 |
|
DYS395Sb |
0,00025 |
0,0000100 |
100 000 |
5,00 |
|
DYS450 |
0,00029 |
0,0000116 |
86 207 |
5,80 |
|
DYS617 |
0.00050 |
0,0000200 |
50 000 |
10,00 |
|
DYS531 |
0.00050 |
0,0000200 |
50 000 |
10,00 |
|
DYS640 |
0,00051 |
0,0000204 |
49 020 |
10,20 |
|
DYS392 |
0,00052 |
0,0000208 |
48 077 |
10,40 |
|
DYS568 |
0,00053 |
0,0000212 |
47 170 |
10,60 |
|
DYS395Sa |
0,00053 |
0,0000212 |
47 170 |
10,60 |
|
DYS438 |
0.00055 |
0,0000220 |
45 454 |
11,00 |
|
Razem 120 mutacji na 500.000 lat, tj. 1 mut. na 4167 lat |
||||
Uwaga. Obliczanie czasu wymaga umiejętności liczenia mutacji
i genetycznego dystansu do najbliższego wspólnego przodka – MRCA.
Attention. Time calculation requires counting skills mutations
and genetic distance to the nearest common ancestor – MRCA.
4a. Tabela
21 powolnych markerów
i ich pełny czas mutacji STR
w zakresie 67 markerowego haplotypu. Wykres wykładniczy.
Podstawą obliczeń w tabeli jest statystycznie określona obecność jednej mutacji w najbardziej wolno mutującym markerze, DYS425 = 500000 lat, a tym samym i w pozostałych markerach tego haplotypu..
Table of mutations time
in 21 slow markers
in scope of 67-markers haplotype STR. Exponential graph.
The basis of calculation in the table is statistically determined the
presence of one mutation
in the most slow mutating marker, DYS425 = 500,000 years, and thus, and
other markers of this haplotype.
|
Obliczanie pełnego czasu mutacji ( PCM ) |
||
|
GD od |
Czas widocznych mutacji
i jego wykładniczy mnożnik, |
Lata od |
|
1 |
4167 x 1 |
4 167 |
|
2 |
8334 x 1,0911 |
9 093 |
|
3 |
12501 x 1,1904 |
14 881 |
|
4 |
16668 x 1,2988 |
21 648 |
|
5 |
20835 x 1,4171 |
29 525 |
|
6 |
25002 x 1,5461 |
38 656 |
|
7 |
29169 x 1,6869 |
49 205 |
|
8 |
33336 x 1,8405 |
61 355 |
|
9 |
37503 x 2,0080 |
75 306 |
|
10 |
41670 x 2,1909 |
91 295 |
|
11 |
45837 x 2,3904 |
109 569 |
|
12 |
50004 x 2,6080 |
130 410 |
|
13 |
54171 x 2,8455 |
154 144 |
|
14 |
58338 x 3,1046 |
181 116 |
|
15 |
62505 x 3,3873 |
211 723 |
|
16 |
66672 x 3,6957 |
246 400 |
|
17 |
70839 x 4,0322 |
285 637 |
|
18 |
75006 x 4,3993 |
329 974 |
|
19 |
79173 x 4,8000 |
380 030 |
|
20 |
83340 x 5,2370 |
436 452 |
|
21 |
87507 x 5,71383 |
500 000 |
Ulepszono / Improved / 10.08.2012, Stanisław Pietrzak
.......................................................................................
...............................................................................
.......................................................................................
...............................................................................
5.
Sposób liczenia mutacji w haplotypie,
genetycznego
dystansu i czasu do najbliższego wspólnego przodka (TMRCA)
C. D. N.
6. Inny sposób obliczania czasu haplogrupy
- przy pomocy mutacji SNP
|
Powyższa tablica PCM haplotypu 67-markerowego ułatwia także, jak żadna
inna, datowanie
odległości czasowej od "chromosomalnego Adama"
do dziś. Na przykład porównanie haplotypu Dorsey'a z haplogrupy
A0 i haplotypów klanu Boyd z haplogrupy A1 daje wynik 55 GD do
wspólnego przodka, Adama, czyli 136.000 lat. (Na takie samo datowanie niespodziewanie wskazały świeżo opublikowane (maj 2012) wyniki dokładnych badań genealogii genetycznej i drzewa filogenetycznego ludzkich szczepów bakterii żołądkowej helikobakter pylori. Wynik identyczny jak powyższy: około 88.000-116.000 lat) Potwierdzenie daje także moja pierwsza próba datowania Adama na podstawie liczby mutacji SNP. Mianowicie poniżej A* (Adam), czyli od haplogrup A1 do R włącznie, w ramach testowania w programie WTY, znajdujemy na dziś 63 mutacje typu SNP.
W najdokładniej testowanej haplogrupie R1b od jej początku,
czyli R-M343 do dzisiejszych ludzi, w kilku
przykładowych gałęziach znajdujemy następujące liczby SNP:
T. Karafet haplogrupie R1 wyliczyła 18.500
lat, co oznacza 1 SNP na 617 lat. Wynik powyższy, obliczony tylko dla 163 SNP, na pewno ulegnie zmianie po uwzględnieniu nowych mutacji STR i SNP niewielkiej grupy Pigmejów i Buszmenów (tzw. klan Perry'ego), której testowanie (z naszej inicjatywy i głównie naszymi nakładami finansowymi !) przeprowadzono w Stanach Zjednoczonych w FTDNA i aktualnie (miesiące IV-VIII) trwa opracowywanie wyników na Uniwersytecie Arizona i Uniwersytecie Houston.
................................
;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;
Nadto oczekujemy na nowe dane w sprawie czasu rozejścia się
człowieka z istotami człekokształtnymi a także w sprawie tem,pa
mutacji SNP w YDNA.
Oto niektóre najnowsze dane w ważnej publikacji z 13 lipca 2012: ;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;
Zobacz
haplotypy Y-DNA haplogrupy A. Wymagają bardziej dokładnego
testowania.
Zob. polscy
misjonarze wśród Pigmejów Batwa-Twa.
;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;;
6a. Pierwsza rozpoznana
mutacja Adama,
Wyjaśnienie dra T. Krahna z genetycznego
laboratorium Houston na RootsWeb |
(Metoda obliczania TMRCA, stosowana zwłaszcza przez genetykę populacyjną, polega na wykorzystywaniu różnorodności, czyli sumy mutacji w całej grupie. Jest ona jednak bardzo mało wiarygodna, gdyż w dwóch grupach, powstałych w tym samym czasie i liczących np. po 100 osób, ilość mutacji może się bardzo różnić, w zależności od tego, kiedy grupa się rozrosła np. z kilku do stu osób. Jeśli wnet po powstaniu, czyli po TMRCA, to mutacji będzie bardzo dużo. Jeśli niedawno temu, to mutacji, przy tej samej liczbie osób, będzie bardzo mało. Ale genetyk, nieznający historii tej grupy, na podstawie niejednakowej różnorodności genetycznej, przypisze im bardzo różniące się czasy).
7.
Dyskusje i
uwagi
Pouczające jest porównanie czasu mutacji w standardowym, 67-markerowym haplotypie, według stawek mutacji i obliczeń czasu przez rosyjską Akademię Genealogii DNA A. Klosowa, z doliczonym czasem mutacji zwrotnych,oraz naszej tablicy tzw. pełnego czasu mutacji (PCM). Podane wyniki - w latach.
| Liczba mutacji |
Tablica AG DNA |
Tablica PCM / FMT |
|
1 |
208 |
111 |
Dlatego genealodzy rosyjscy (w osobie A. Klosowa),. widząc niesprawność swojej tablicy obliczania czasu, na forum RootsWeb stwierdzają, że ich tablica ma przeznaczenie tylko do dystansu do około 15 tysięcy lat. Dla dalszego czasu radzą oni używać ich tabelę 22-markerową. Ta wykazuje jednak identyczne mankamenty, co poprzednia, tylko że na innym dystansie czasu.
Zauważamy, że tabela PCM, uwzględniając rachubę wszystkich mutacji powtórnych (niewidocznych), które znacznie opóźniają pojawianie się bardziej licznych niż kilka-kilkanaście mutacji prostych (widocznych), umożliwia datowanie haplotypów bardzo dawnych, sięgających Adama, przodka dzisiaj żyjących ludzi - około 142 tysiące lat (jak rachował Cruciani et al. 2011), a średnio i właściwie 103.000, jak tu rachujemy.
W Y-DNA człowieka nie zauważamy więcej mutacji prostych względem MRCA niż 50 - 60, np. dla haplogrupy A (chromosomalny Adam). Reszta czasu zakodowana jest w ilości mutacji powtórnych (niewidocznych). Tabela PCM uwzględnia je i wprowadza w ogólną liczbę mutacji.
Tablica PCM czyni zbędnymi starania dla wypracowania "ewolucyjnego tempa mutacji", jak czyni Zhivotovsky dla starszych ponad 5000 lat haplotypów. Podobnie niepotrzebne jest tworzenie osobnej tablicy mutacji na podstawie 22 markerów powoli mutujących, jak czyni AKlyosov dla obliczania czasu haplogrup i haplotypów sprzed 20000 lat, dokąd podstawowa tabela zasadniczo nie sięga. Na powyższym zestawieniu widać potrzebę owej dodatkowej tabeli dla naprawienia olbrzymich rozbieżności między rezultatami podstawowej tabeli AK a faktycznym czasem mutacji.
Uwaga. Nie ma sensu i jest
zdecydowanie błędne rachowanie czasu według odległości genetycznej dwóch
haplotypów (dwóch osób). Spotkane np. GD=100 między dwoma haplotypami należy
najpierw podzielić przez dwa, aby uzyskać GD i czas
wspólnego przodka MRCA.
W ogóle problemem nadal do
rozwiązania jest sposób liczenia liczby mutacji i dystansu genetycznego
między dwoma haplotypami do współnego przodka.
E-mail:
pietrzakstan@poczta.onet.pl
Wszelkie uwagi dla mnie proszę
przekazać meilowemu gołąbkowi:

Dziękuję,
już dziś przekażę Adresatowi
|
DZIĘKUJĘ! |
|
GOŚCIEM |
 Po testach grupy Perry'ego, odkryciu i opublikowaniu kolejnych
licznych mutacji SNP, ogólna ich liczba w genealogii
najdokładniej przetestowanych próbek haplogrupy R1b wynosi około
203.
Po testach grupy Perry'ego, odkryciu i opublikowaniu kolejnych
licznych mutacji SNP, ogólna ich liczba w genealogii
najdokładniej przetestowanych próbek haplogrupy R1b wynosi około
203.