|
6 .
tekst zaktualizowany
28.10.2015
Genetyczne pochodzenie Pietrzaków
od pierwszego mężczyzny - genetycznego Adama (L74).
Droga naszych przodków od biblijnego Raju (w Afryce) do Łukowicy !
Nasza najmłodsza
znana mutacja
pod Z280:
YP380;
Otwórz stronę pdf. Po powiększeniu zobaczysz drzewo
wielkich genetycznych rodów świata, wywodzących się od pionu
"Y-Adam-Pietrzakowie" z symbolami YP343-YP380. Na tle całego drzewa
zobaczysz miejsce naszego rodu.
http://www.tropie.tarnow.opoka.org.pl/Genetyczne.rody.Y-DNA.w.swiecie.pdf
Genetyczna genealogia Pietrzaków,
zapisana w ojcowskim Y-DNA
(Mój pogląd na świat:
Kreacjonizm
ewolucjonistyczny
The evolutionistic
creationism
)
.
|
 |
MAPA naszego ojcowskiego rodowodu od Afryki do
Europy
Od A i B w Afryce, przez CF i F na Bliskim Wschodzie,
IJK, K, M i P w Azji Środkowej,
oraz R (R1, R1a) na północ od Kaukazu
(według pracy
Chiaroni et al. 2009). |
Naszą najstarszą
genealogię, tam gdzie nie sięgają zapisy ksiąg metrykalnych, inne dokumenty
i tradycje rodowe, odkrywa GENEALOGIA GENETYCZNA, zapisana zwłaszcza w
męskim/ojcowskim Y-DNA - jeśli chodzi o genealogię mężczyzn/ojców oraz w
mitochondriach mtDNA - jeśli chodzi o genealogię matczyną mężczyzn i kobiet.
Męski CHROMOSOM Y-DNA ma to do
siebie, że jest przekazywany tylko męskim potomkom i wiernie, to znaczy bez
żadnej rekombinacji z żeńskim X-DNA. Dziedziczone są przy tym wszystkie
powstałe uprzednio w Y-DNA mutacje w pełnym zestawie, o ile trakcie
zapłodnienia, na skutek błędu w kopiowaniu, nie dołączy kolejna, nowa. Do
mutacji dochodzi w różnych miejscach Y-DNA; w jednych są bardzo rzadkie,
zdarzają się zasadniczo tylko raz i tylko u jednego mężczyzny. W innych
segmentach zachodzą często, mogą więc zdarzyć się takie same u wielu mężczyzn
równocześnie, niepowiązanych żadnymi nićmi pokrewieństwa.
Mutacje rzadkie (zwane
wielkimi) wyróżniają haplogrupy, a częste (zwane małymi) wyróżniają haplotypy.
HAPLOGRUPA (od gr. 'haplos'-
pojedynczy, jednakowy) oznacza grupę ludzi, wyróżniających się
identyczną mutacją w SNP (single nucleotide polymorphism, w skrócie: snip) męskiego Y-DNA,
wiernie dziedziczoną od wspólnego przodka, w którym dokonała się owa
określona mutacja. Takich mutacji rozpoznano do dziś około tysiąca, a ich
rejestr znajduje się na stronie
ISOGG (na DNA naszego przodka Macieja takich mutacji na drodze dziedziczenia nagromadziło się około 102).
To dziedziczenie całego zestawu dawnych mutacji pomaga zestawić je w
formie drzewa genealogicznego mutacji i zarazem ludzkości, które też
prezentuje na kolejnej stronie instytucja
ISOGG.
Nasze korzenie, jak i korzenie wszystkich współczesnych ludzi,
jak na to wskazuje archeologia i drzewo genealogiczne mutacji SNP, sięgają Afryki Środkowo-Zachodniej.
Tam miał żyć pierwszy mężczyzna, nasz praojciec z gatunku
Anatomicznie Współczesny Człowiek (AMH).
|
Był to naprawdę współczesny człowiek, który ponad 100
tysięcy lat temu, jak wskazują np. archeologiczne pozostałości w
jaskini Blombos w południowej Afryce, tworzył z
minerałów i składników
przyrodniczych doskonałą farbę koloru ochry, w tym
miejscu na stronie celowo przeze mnie zaprezentowanej,
doskonały i silny klej do
przylepiania grotów do strzał i niegorsze
ozdoby oraz narzędzia kamienne i kościane i zostawił nam
pierwsze jaskiniowe naskalne malowidła i ryte ornamenty. |
Grupę genetyczną Y-DNA,
najbliższą naszemu wspólnemu przodkowi, genetyka oznaczyła symbolem
A(dam), od biblijnego
imienia pierwszego człowieka. Kolejne litery alfabetu od A do S oznaczają
kolejne haplogrupy, określane symbolami kolejnych liter i cyfr na potrzeby budowy drzewa genealogicznego
ludzkości. Haplogrupa R, zwłaszcza jej podgrupa R1a i jej podgrupy, dotyczy
rdzennych Słowian, a więc - jak się okazało - i nas, Pietrzaków.
HAPLOTYP zaś to pewien zestaw
częstych mutacji STR (short tandem repeats), dokonanych w rejonie
mikrosatelitarnym Y-DNA. Mutacje te rozpoznawane są po zmiennej liczbie
powtórzonych tandemów (sekwencji) nukleotydów. Liczba powtórzeń w danym
miejscu rejonu mikrosatelitarnego nazywa sie allelem. Te właśnie allele,
rozpoznane w 25, 37 lub 67 miejscach (loci, markery), stanowią o
indywidualnym haplotypie każdego mężczyzny. Przykład takiego
haplotypu, w opracowaniu FamilyTreeDNA dla Macieja Pietrzaka, podany jest na samym dole tej strony.
Jednakowe haplotypy, zwłaszcza
dłuższe, 67-markerowe, mogą powtarzać się zasadniczo tylko u najbliższych
krewnych. Ich odrębności więc mogą być użyteczne do oceniania dystansu genetycznego (GD), czyli
stopnia pokrewieństwa dwóch osób (choć nigdy nie ze stuprocentową
pewnością!), albo do wyróżniania klastrów, czyli większych krewniaczych
grup ludzi, tam, gdzie haplogrupy są zbyt wielkie, jak np. euro-azjatycka haplogrupa
R1a1a1, licząca obecnie może około 200 milionów ludzi w Europie i Azji.
(Allele mogą
posłużyć też do
obliczania wieku populacji danej haplogrupy lub haplotypu (choć też nigdy ze
stuprocentową pewnością). Czas pierwszego wspólnego przodka dla dwóch i
więcej haplotypów oblicza się i sprawdza kilkoma metodami równocześnie, ale
zawsze na podstawie liczby mutacji w poszczególnych haplotypach. Do mutacji
bowiem dochodzi przypadkowo, ale przy większej ich liczbie metodami
statystycznymi ustalono współczynnik czasowy ich występowania. Np. gdy
między dwoma 67-markerowymi haplotypami brak mutacji to znaczy, że z
95-procentową pewnością mogą mieć wspólnego przodka (MRCA) od 1-6 pokoleń,
czyli do 150 lat, licząc po 25 lat na pokolenie. Gdy zaś między nimi
zachodzi jedna mutacja, to można mieć taką samą 95% pewność, że ich
MRCA żył w
czasie od jednego do 9 pokoleń, czyli do 225 lat. Przy wielkiej liczbie mutacji współczynnik ten stabilizuje się w
środku tego zakresu, czyli 1 mutacja w 67-markerowym haplotypie może być
liczona na około 112 lat).
KOD Y-DNA chromosomalnego Adama (Y-Adama)
-
To tysiące mutacji SNP (snip) ojca całej ludzkości,
chromosomalnego Adama, które oddzialają go od szympansa.
Wśród
nich jako pierwszy został rozpoznany snip L74;
jest on ziedziczony od chromosomalnego Adama przez wszystkich mężczyzn na świecie.
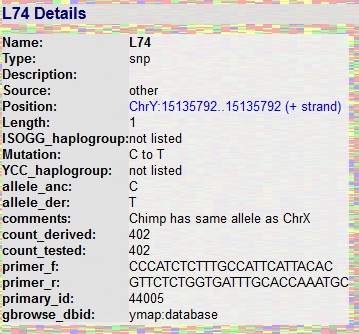
Snip L74 nie został wprowadzony na listę mutacji ("non listed")
ISOGG, gdyż rejestruje on tylko te mutacje,
którymi tylko grupy ludzkie różniż się między sobą.
|
Członkowie rodu łukowickich Pietrzaków
uczestniczą
czynnie w naukowych i
laboratoryjnych poszukiwaniach miejsca
"chromosomalnego Adama" i
jego najbliższych potomków,
co ujawnia
A-haplogroup
FTDNA Project i
liczne strony internetowe.
(Mój
pogląd na świat:
Kreacjonizm ewolucjonistyczny
) |
Oto w kierowanym przez B.
Schrack projekcie
badań haplogrupy A (zob.
Y-haplogroup A Family Tree DNA Project
oraz Haplogroup A, the Root of the
Tree of our Fathers
), przy naszym decydującym udziałe, o czym można czytać na stronie
Y-Adam, doszło do ustalenia
najstarszych gałęzi ludzkiego rodu i miejsce ich powstania.
Przedstawia to
poniższa mapa:
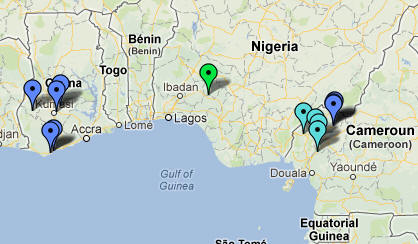
Pineska zielona oznacza archeologiczne szczątki praczłowieka z
Iwo Eleru.
Kolor jasnoniebieski - to najstarsza haplogrupa A00 w plemieniu Bangwa i
Nkongho, w Południowo-zachodnim Kamerunie.
Kolor ciemnoniebieski - to haplogrupa A0 w plemieniu Nso w powiecie Kumbo w
Północno-zachodnim Kamerunie
i nieco młodsze w Ghanie.
Teraz - po kolei haplogrupy
potomków ADAMA na genetycznej drodze do PIETRZAKÓW
(przy nazwie haplogrupy wymienia się spośród wielu mutacji zasadniczo tylko
jedną, jakby definiującą dany ród.
1. Hapologrupa A
Na drodze zestawienia mutacji STR niektórych ludzi z haplogrupy A,
zwłaszcza Pigmejów i Buszmenów, można obliczyć genetyczny dystans (GD) między
nimi a ich najbliższym wspólnym przodkiem (MRCA), czyli owym
chromosomalnym Adamem. Wyniki badań F. Crucianiego i zespołu (2009)
oraz kolejne późniejsze testy Jenesa i Dorseya
potwierdziły datowanie Y-Adama, chromosomalnego przodka współcześnie
żyjących ludzi, na około 142.000 lat.
Ale ostatnie dokonane
także przy naszym udziale testowanie Perry'ego i jego genetycznych krewnych
z Kamerunu wskazały na daleko wcześniejsze datowanie
wspólnego przodka wszystkich współcześnie żyjących ludzi. Wzrost liczby
mutacji (SNP) między Y-Adamem a dzisiejszymi ludźmi pozwolił na datowanie
tego wspólnego przodka na 338.000 lat (Hammer
2012; Mendez 2013), ale przy
założeniu że jedno pokolenie liczy przeciętnie 30 lat. Po przeliczeniu
pokolenia na 25 lat (zobacz) oraz
po zastosowaniu poprawionego tempa mutacji w chromosomie Y (według Fu et al.
2014) trzeba
mówić, że: Y-Adam żył około 250.000 lat temu.
|
Najbliższy wspólny
ojciec dzisiejszej ludzkości,
czyli tzw. chromosomalny Adam (Y-Adam),
jak wskazuje aktualna
liczba mutacji (SNP) w jednej linii od hg. A0T do R
(aktualnie, po październiku 2013, około 2100 SNP)
lub genetyczny dystans między człowiekiem a szympansem
i inne badania, np. Wei et al. 2013, Shi et al. 2013, Fu
et al. 2014
Y-Adam żył
około
250 tys. lat temu
Znaczy to także, że każdy z nas
od czasów owego Adama
ma ponad 10 tysięcy praojców i
pramatek.
Podobne wyniki dają badania mitochondrialnego mtDNA
matek. |
Dwie grupy najbliższych potomków
Adama, czyli haplogrupy A
- A00 z mutacjami grupy
L1086 i
in. - niektóre plemiona, zwłaszcza Bangwa/Nweh i Mbo/Samba
w Południowo-zachodnim Kamerunie.
- A0 z mutacjami grupy L1085 i in. - zwłaszcza Nso w Północno-zachodnim Kamerunie
i w Ghanie
- A1 z mutacjami P305,V161 i in., w
Afryce, dziedziczonymi przez wszystkich pozaafrykańskich mężczyzn.
(zob.
drzewo genealogiczn potomków Adama, sporządzone po wykonaniu
testowania WTY czterech Afrykańczyków)
Zob. najnowszą tablicę czasu głównych rodów (haplogrup) w pionie drzewa od
Y-Adma do nas (XI 2014)
http://www.tropie.tarnow.opoka.org.pl/images/czas_hg.jpg
2. Haplogrupa BT - wypływająca z haplogrupa A jako
A1b2; określa ją mutacja M42 i inne,
która Wyloniła się się u jednego z naszych
praojców około 135.000 lat termu, zapewne już we Wschodniej Afryce, a
rozgałęziła około 98.000 lat temu na afrykańską B i pozaafrykańską CT. Dziedziczona
jest przez wszystkich mężczyzn
poza Afryką, a więc także i wszystkich kolejnych przodków Macieja Pietrzaka w
linii prostej. (Uwaga. Do naszych badań na
przełomie 2011/2012 haplogrupie BT przypisywano
starszeństwo i zupełną odrębność od afrykańskiej hgA. W tej grupie widziano
przodka całej ludzkości, z wykluczeniem afrykańskiej haplogrupy A).
3. Haplogrupa CT - z niej około 70.000 lat temu powstała
w Palestynie haplogrupa E (która częściowo przeszła do południowej Europy, a
częściowo do Afryki, gdzie regionalnie rozwinęła się do 100% miejscowej
ludności; i wschodnioazjatyckie (japońskie) D i Mężczyźni z haplogrupą
D żyją np. w Japonii, a C żyją
dziś głównie na południowych obrzeżach Azji, od Arabii przez Indie i
Indonezję - po Australię (Aborygeni), wyspy Polinezji, kraje Azji wschodniej i
Indian w Ameryce Północnej i Południowej. Haplogrupa C jest gałęzią boczną -
nie było wśród niej naszych przodków.
3a. Haplogrupa CF. Niewiele później po
roku 70.000 lat temu wydała ród C, żyjący głównie w Australii i Azji
Południowo-Wschodniej (Chiny)
4. Haplogrupa F - z mutacją M89, wydzieliła się u jednego z
kolejnych naszych wspólnych przodków z populacji CF, po mutacji M89, do
której doszło gdzieś w Azji Środkowej lub południowej.
5. Haplogrupa IJK - cechuje kolejnego naszego wspólnego przodka, z
którego powstały trzy populacje: staroeuropejska I1 i I2 (18% ludności
polodowcowej Europy), bliskowschodnia J1
i J2 (jako gałęzie boczne) oraz nasza gałąź euroazjatycka K .
6. Haplogrupa K - z mutacją M9, pod kolejną mutacją,
K2-M526, rozdzieliła się, gdzieś w
rejonie dawnego Sundlandu (Azja Poludniowo-Wschodnia), około
50.000 lat temu, na wiele podgałęzi, w
tym najważniejszą dla nas K2b=MP, żyjącą na
byłym Sundalandzie (przed jego zalewem), gdzie dziś tylko Wyspy Sundajskie
(m.in. państwo Indonezja) (inną krótką podgałąź wykazały archeologiczne
szczątki tzw. "Człowieka z Ust'-Ishim" nad rzeką Irtysz na Syberii a roku
45.000 lat temu, o czym informacja jest na stronie:
http://www.tropie.tarnow.opoka.org.pl/polacy.htm
7. Haplogrupy N, O i P - to potomkowie haplogrupy K2. N to haplogrupa
chińska, mongolska, syberyjska, uralska północnorosyjska i skandynawska (tu
jako Ugrofinowie i Samowie). Haplogrupa O, upowszechniona w Południowo-Wschodniej Azji
(Chiny)
i na wielkich wyspach
zachodniego Pacyfiku, i wreszcie "nasza" haplogrupa P., bo jej
"założyciel" był jednym z naszych praojców.
8. Haplogrupa P - z mutacją M45,
rozgałęziła się około 36.000 lat temu gdzieś w Azji środkowo-wschodniej lub
poludniowej. Z niej
około 35.000 lat temu wydzielił się przodek haplogrupy Q, która dała początek niektórym ludom
ałtajskim, północno-wschodniosyberyjskim i - głównie - Indianom amerykańskim
(obok małej garstki C, już tam może wcześniej przybyłej). Z haplogrupy P wydzielił się też przodek naszej,
eurazyjskiej haplogrupy R.
9. Haplogrupa R - powstała dzięki mutacji M207 u naszego praojca około
35.000 lat temu gdzież może w Azji Południowo-wschodniej. W niej
około 33.000 lat temu wydzieliła się haplogrupa R2 głównie dla Indii oraz ważna dla nas, nasza ojcowska haplogrupa R1-M173.
10. Haplogrupa R1-M173 - powstała około
33.000 lat temu. Miejscem powstania była może Azja Środkowa, zwłaszcza rejon
Ałtaju. Tam właśnie (pod Bajkałem) archeolodzy znaleźli szczątki tzw.
"chłopca z Mal'ty", którego DNA ujawniło inną krótką boczną gałąź haplogrupy
R.
Jeden z
potomków tej haplogrupy wydzielił się około 29.000 lat temu z mutacją
M343, dając początek haplogrupie R1b, później (już w Europie) italo-celtyckiej. Stał się w ten
sposób ojcem
głownie zachodnich Europejczyków - dziś około 110 milionów ludzi. Druga gałąź wyłoniona spod R1
około 29.000 lat temu to haplolgrupa R1a; w niej pojawił sie i
nasz ród Pietrzaków.
11. Haplogrupa R1a wydzieliła się
dzięki mutacjom M420 i kilku innym zapewne około
29.000 lat temu, prawdopodobnie w rejonie
Azji Środkowej, gdzie też żyła jej ojcowska haplogrupa R1. Mutacja ta jest dziedziczona przez
wszystkich męskich jej potomków do dziś.
13. Haplogrupa R1a1a z mutacjami
M17 i M198 -
wydzieliła się 17 tysięcy lat temu lat temu, zapewne gdzieś w Azji.
14.
Haplogrupa R1a1a1 z mutacją
M417.
Wyłoniła sie ona około 8,500 lat przed Chr., prawdopodobnie w "drodze" w
kierunku ziem polskich (Kujawy?), gdzie żyła prawdopodobnie w
archeologicznej kulturze janisławicko-bugoostrowskiej, a potem amfor
kulistych, które ewoluowała na Kujawach. Około 5.400 lat przed Chr. powstają jej dwie podgrupy:
północno-zachodnioeuropejska CTS4385
oraz euroazjatycka Z645.
Zob. Miejsce M417 na drzewie R1a i jego gałęzie.
https://upload.wikimedia.org/wikipedia/commons/d/d4/R1a._Info-19.05.2014.jpg
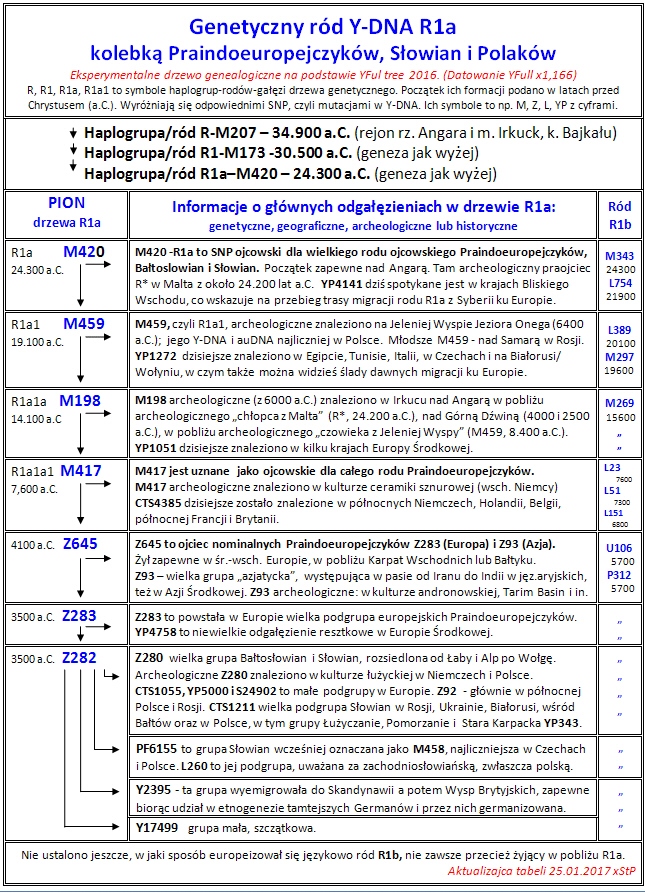
15. Mutacja Z645 rozwijająca się w
latach 5.400-4.500 lat przed Chr. rozgałęziła się na potężną gałąź europejską
Z283 i jeszcze większą, zasadniczo azjatycką Z93.
16. Mutacja Z93. Upowszechniła się ona głównie w Azji
Środkowej (np. Kirgizja) oraz, pod słynną nazwą Ariowie, w Iranie i na półwyspie Indyjskim;
tam zanieśli azjatycką wersję języków "indoeuropejskich" i stworzyli tzw.
święte księgi Wedy i Awesty. Dziś tę haplogrupę rozpoznaje się u
około 100
milionów męskich mieszkańców Półwyspu Indyjskiego (ok. 16%), podobnie w
Iranie i Pakistanie (ok. 16%-25%)
17. Pod mutacją Z283 i kolejnej - Z282,
której rozgałęzienie obecnie jest datowan3 na
4.225
lat przed Chr., wyłoniły się trzy wielkie gałęzie ze swoimi
mutacjami; można im przypisać
tworzenie rozległego w Europie archeologicznej kultury amfor kulistych (od
około 4.000 lat przed Chr.) i horyzontu kultur ceramiki
sznurowej (ang. Coreded Ware culture), rozwijającego się od około 2900 do 2200
lat przed Chr.
(http://en.wikipedia.org/wiki/Corded_Ware_culture):
a) mutacja Z284 i pod nią głównie L664,
mniejszościowa, żyjąca dziś głównie w Europie północno-zachodniej
(Skandynawia, Wyspy Brytyjskie),
b) mutacja M458, nazwana przez
Underhilla środkowoeuropejską. Wyłoniła się,
jak cała ta trójka, około 4.000 lat przed Chr., gdzieś
może w rejonieWisła-Odra, obejmująca dziś może kilkadziesiąt milionów ludzi od Bawarii przez Czechy i Węgry po Zakarpacie,
Ukrainę i
Polskę. W niej wnet potem wydzieliła się gałąź
zachodniosłowiańska (głównie w Polsce i sąsiednich krajach) wyróżniająca się mutacją
L260.
c) mutacja Z280, powstała tak samo około
3.700 przed Chr. Tworzy ona dziś
bardzo wielką populację w Europie Środkowo-Wschodniej, prawie wyłącznie
wśród Słowian i Bałtów.
Oto dokument naszej mutacji Z280, oznaczającej początek wielkiego rodu
bałtosłowiańskiego:

Miejsce tej mutacji na chromosomie Y oznaczone jest liczbą kolejną
Chry:6289881.
Mutacja ta to zmiana C->T, czyli zasady cytozyny w tyminę.
Zobacz drzewo genogenealogiczne tej olbrzymiej grupy Z280.
Stan badań z 27 grudnia 2014
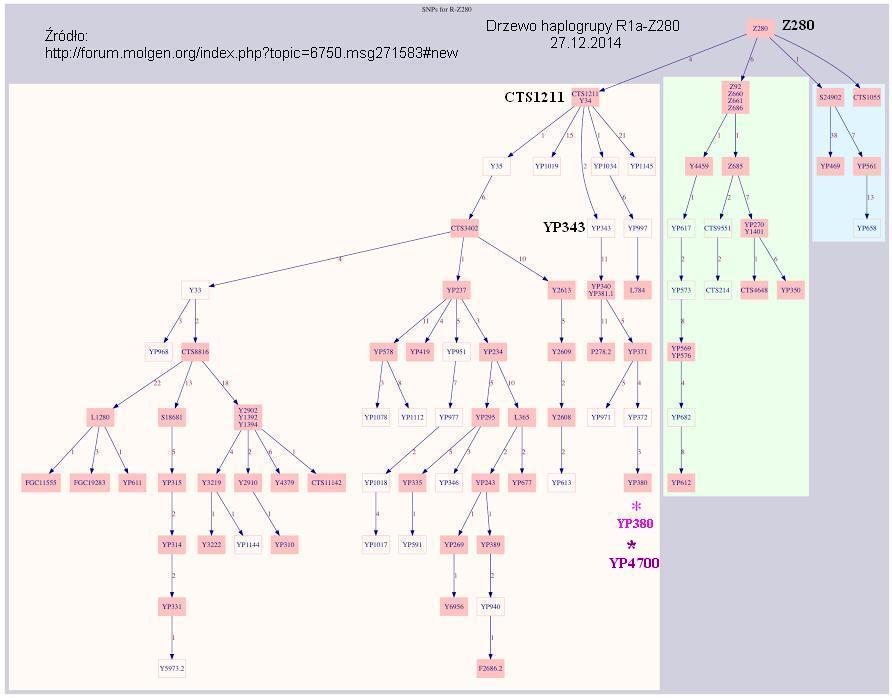
18. Pod mutacją
Z280 wyłoniły się cztery gałęzie: niezbyt liczna
Z92, która rozprzestrzeniła sie
głównie w rejonie Południowego Bałtyku i wśród Słowian Wschodnich, dwie
jeszcze mniejsze i mało znane, oraz
czwarta, bardzo liczna - CTS1211.
19. CTS1211 uległa podziałowi na pięć
gałęzi, w tym Y35 (CTS3402) dała początek wielkiej grupie środkowo-wschodniej, z
wielką podgrupą zwaną Wołga-Karpaty oraz gałęzią karpacką YP343,
naszą, niezbyt liczną.
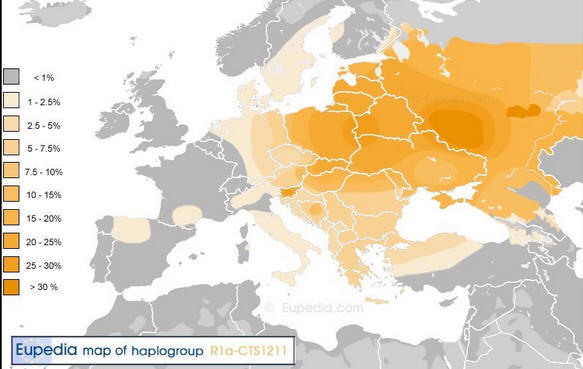
20. Haplogrupa
YP343, tzw.
karpacka (Carpathian) -
2.300
przed Chr.
Aktualne miejsce na drzewie Y-DNA (haplogrupa) oznaczono symbolem
R1a-YP343
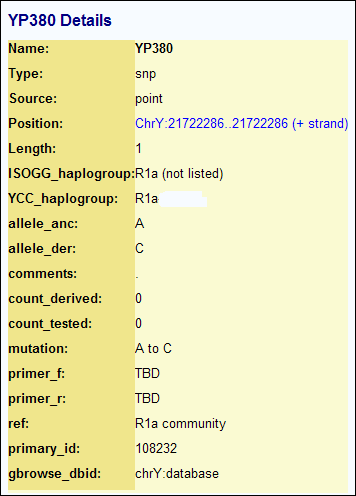
Ostatni, ustalony SNP w rodzie Pietrzaków;
YP380, datowany na rok około
500-900 r. AD,
Jest
wspólny z rodem Rauch, żyjącym nad
Solawą w Niemczech (Serbo-Łyżyczanie?) rodem
Stańków ze Śląska Cieszyńskiego i
rodem Galovič
ze Spiszu Slowackiego. Nowsze nasze mutacje to sześć z serii
YP4700...5.
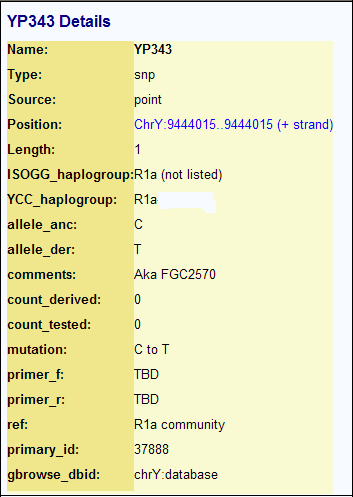
Oto "metryka" mutacji 343
i 380 z pracowni FGC:
Wyłoniła się ona, podobnie jak CTS3402, około
2,500 r. przed Chr. (ang. ybJC) Gałąź ta
została rozpoznana w testowaniu Big Y-DNA, w kwietniu 2014 r. Rozpoznano
wtedy ponad 30 mutacji typu SNP, które otrzymały oznaczenia
YP343...345 oraz YP371...381.
Czego dowiedzieliśmy się o
ludności
gałęzi YP343? Pojawiła się ona około 2,300 r. przed Chr. Jej
podgałęzie rozprzestrzeniły się dość dość szeroko w Europie, zwłaszcza środkowej, choć
nawiązują do rejonu Karpat.
Ludność ta, wraz z całą grupą Z280 i M458, a może i
Z284, zapewne uczestniczyła w szerokim upowszechnianiu
się późniejszej fazy archeologicznej kultury ceramiki
sznurowej, a także w tworzeniu i rozwijaniu kultury epoki brązu, zwanej Otomani-Fuzesabony w Kotlinie Karpackiej
oraz u nas, po północnej stronie
Karpat (np. w Maszkowicach, o czym niżej).
Na powyższym dokumencie określono nazwę mutacji: YP343 i numer miejsce na
chromosomie Y: Chry:9444015
.
........................................................... ......................................................
*Miejsce
mutacji YP343 i YP380 na gałęzi Z280
(starsza wersja drzewa)
Podstawowa gałąź ludów słowiańskich, to Z280
(obok mniej licznej, środkowo-zachodniosłowiańskiej M458)
Wydzielenie się gałęzi Z280 - około 3900 lat przed Chr.
Niżej - stan gałęzi Z280 i miejsce Starej karpackiej (Old Carpathian)
podgałęzi, YP343, oraz jej gałązek.
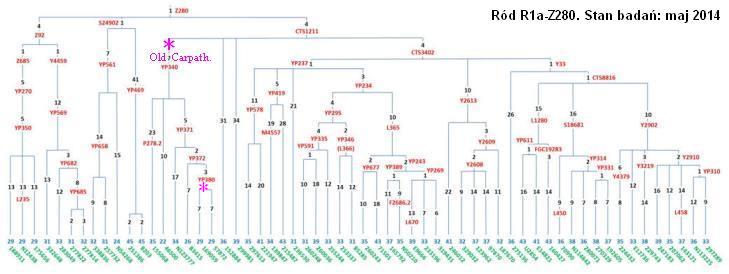
Opis diagramu
1. Cyfry koloru czarnego oznaczają liczbę mutacji typu SNP, ujawnionych w testowaniu
BIG Y-DNA. W niektórych punktach znajduje się wpisany także czas haplogrupy, który
oznacza jej podział na przeważnie dwie podgałęzie, a zarazem
wyłonienie się nowych haplogrup. W zależności od liczby SNP w haplogrupie,
dystans między wyłonieniem się a faktycznym czasem (rozgałęzieniem)
haplogrupy, równa się wielokrotności czasu jednej mutacji.
Także kolorem czarnym: Stosownie do liczby SNP obliczono czas haplogrupu
2. Symbole i cyfry czerwone oznaczają oddzielne haplogrupy, czyli
grupy ludzi odznaczających się
identyczną jedną lub zespołem identycznych
mutacji typu SNP.
3. Gwiazdka * to gałąź Stara karpacka
4. Niebieskie
cyfry (na dole)
oznaczają sumę mutacji na całej linii od
pierwszego wspólnego przodka, czyli Z282.
5. Zielone cyfry (na dole)
oznaczają laboratoryjne oznaczenia każdej testowanej próbki, tworzące
własne gałązki.
Nasza
jest oznaczona: 169059. Oto obraz gałęzi Starej karpackiej z naszą
gałązką (stan badań: październik 2014)
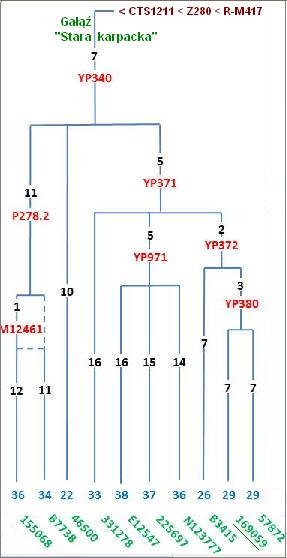
Gałąź Stara karpacka na mapie, z jej młodszymi rozgałęzieniami:
http://i1127.photobucket.com/albums/l625/ft-d/5Q.jpg
lub w zestawie:
http://www.familytreedna.com/public/R1a/default.aspx?section=results
Po powiększeniu powyższej
mapy widzimy zagęszczenie w małopolskich Karpatach, gdzie znaleziono
przedstawicieli wszystkich gałęzi Starej karpackiej i jej młodszych
podgałęzi, np. z mutacją P278.
Oto częściowe rozmieszczenie w Europie
i wstępny spis nazwisk tej grupy
(według stanu badań w 2011r. na mapie
Molgen-Semargl.me )
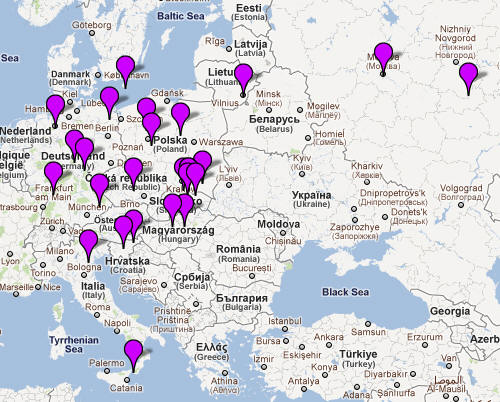
Miejsca części naszych genetycznych krewnych z gałęzi
Starej karpackiej: YP340
oraz jej
podgałęzi na mapie MolGen.
W projekcie 'R1a1a and Subclades' pod oznaczeniem
6. 'Old Carpathian' znajdujemy około 100 testowanych lub wstępnie
zozpoznanych próbek z mutacją YP343 YP343.
|
Zobacz miejsce karpackiej gałęzi YP343 na drzewie
genealogicznym R1a
https://upload.wikimedia.org/wikipedia/commons/d/d4/R1a._Info-19.05.2014.jpg
YP343 „Old Carpathian”
(źródło: R1a and Subclades, 02.01.2016)
https://www.yfull.com/tree/R-YP343/
https://www.familytreedna.com/public/R1a?iframe=yresults, str. 3 |
|
Grupa
"Karpacka" rodu R1a (w niej Pietrzakowie)
HAPLOGRUPY pionu od M417, tj. najbliższego
wspólnego
ojca Praindoeuropejczyków, do naszych YP380/YP4700
Symbol haplogrupy, ilość SNP (.), czas formowania:
M417
(32 snp),
formed 8500-5500 yBP (to
wspólny ojciec Praindoeuropejczyków).
Z645 (8 snp), formed 5500-5000 yBP
Z283
(2 snp),
formed 5000-5000 yBP
Z282 (1 snp), formed 5000-5000 yBP
Z280 (5 snp), formed 5000-4700 yBP
CTS1211 (4 snp), formed 4700-4300 yBP
..........
YP343 (2 snp),
formed 4300-3500 yBP
YP343*-A-x
(Big Y needed)
98340 near Oswiecim, Poland
N38418 Wojciech Bulawa, b.c.1887, Krzyzowa, Poland
YP343*-A-x1
(Big Y or YP340, YP343 needed)
N51999 Jan Lapinski, Janow , Poland
YP3979-A
(13 snp) formed 3500-1300 yBP
316721 Wasyl Naberezny, b. 1894 and d. 1956 ,Poland
362587 Janatik, Otis, Czech Republic
YP3979-B (another
Big Y needed)
33699 Andrew Gazdik, b 1892 and d 1973 ,Slovakia
YP340 (12 snp),
formed 3500-2800 yBP
YP340-x1
(Big Y or Z280 SNP pack needed)
74991 William Runka, b.c.1890, Chortkiv, Ukraine
145439 Michael Hoffman,b.1812,Wischin Hauland,
Prussia,Germany
N17933 Walenty Wytrwal, b.c.1747, Bystrzyca, Podkarpacie,
Poland
224354 (- - - ), Unknown Origin
77875 John Hnatowicz, b.c.1800, Bartne, Poland
199944 Trofim Kavtaev, Russian Federation
266563 Wiktor Solecki b.1820 d.1869 Poland
129045 Franc Pestotnik, b. 1813, Psajnovica, Slovenia
N114571 Symeon(Sam)Starosta,Borschiv,Ukr. d.1950's ,Ukraine
158675 Adam P. Majewski, Dec. 24, 1880, Feb. 8, 1959 Poland
155785 Valentine Janeš or Yones, Tršce, Croatia 1874,
Croatia
117227 Giovanni Battista Donà Italy
YP340*-y
(YP340 not tested, Big Y or Z280 SNP pack needed)
52014 Johann Heinrich Rathke, b 23 Sep 1753, Balin,
Germany
263663 Савелий Борисенко 1918-1968, Russian Federation
360317 Mr. Mihael Hochkraut, b. 1707 and d. 1797
Slovenia
B13734 Saveliy Naumovich Staroselskiy (1800-1870),
Russian Fed.
93464 Vladimirov Russian Federation
YP340*-z
(YP340 not tested, more STRs or Big Y needed)
349258 Michael Albright b.1799 and d. circa 1890 ,
Unknown Origin
220038 Josef Muller b 1851 and d 1906, Austria
442527 Lovász, b 1785, Istensegíts, székely-magyar ,
Hungary
266636 Vladimir I Ladinsky 1918 - 1997 Simbirsk , Russian
Federation
N57404 Budin, beginning of 17.century, Bohemia, Czech
Republic
276761 Galkin Ilushkino Ul'yanovsk region , Russian
Federation
118740 Antal Lovász, b. 1800, Istensegíts, Bukovina
Hungary
79988 Kiss Hungary
N18451 Frank J Zalewski, 1858 - 1941, Goczalki, Poland
322969 Stefan Grabiański, b. 1780 and d. 1827 , Poland
245756 Zoltán Drinóczi, 1954, István Drinóczi, 1931 ,
Hungary
353714 Unknown Origin
293154 Russia, Rostov na Donu, Russian Federation
YP340-A
(YP340 not tested, more STRs and Big Y needed)
N118610 Elinski , Poland
284897 Kefa Stefan Faryna b. 1878 in Ukraine
127575 Ferenc Koródi, b. 1828, Józseffalva, Bukovina ,
Hungary
FGC2555*
326102 Christopher Larsen Snørsrud, c.1716-59, Fet, AKR ,
Norway
FGC2555-A1 (another
Big Y needed)
46500 John Hollister died 1596 Westerleigh, Glos,
England
170611 F05/Frederick McAllister,s.of Thomas, b.1808 PA,
Unknown
FGC2555-A2-x
(FGC2555 not tested, Big Y needed)
N68879 (- - - ) Germany
158625 Hans Hurtzler, About 1520, Uerkheim, Aargau ,
Switzerland
FGC2555-A-x
(FGC2555 not tested, Big Y needed)
308695 England
P278.2*
155068 Julius Johan Baranowski, b. 1879 and d. 1952,
Poland
173752 Nikolae Ivanov Slovtsov, 1782 -1846, Russian
Federation
P278.2*-x (Big Y
needed) [YP1702-]
263026 Ivan Andrusenko, XVIIIc, Vinnitsa r. (Ukraine),
Ukraine
P278.2-x (YP1702
not tested, Big Y needed)
70291 András Kiss b. probably Derecske about 1820, Hungary
221387 Matija Kendjelic, est. 1757 - ?, Croatia
119580 Wawrzyniec Potaczek, b.c.1881, Podobin, Poland
156436 Henning Pedersen, 1656- 1729, Bornholm, DNK,
Denmark
P278.2-x1 (Big Y
and/or 111 STRs needed)
337141 Havrila Vavelyuk, b. circa 1875 , Ukraine
E16966 X, c1700, Hungary
163780 Andreas Kocur,b.1800, (Niewiesze, Silesia?),
Poland
357453 (- - - ) Unknown Origin
79603 Juraj Dadej Slovakia
373501 Mikhail Antonovich Rakovets, b. 1886 , Belarus
N24335 ISMAIL HAKKI KOROGLU, 1892-1944 Turkey
68097 Zaborowo, Poland Poland
141547 Johann Muecke/Mueckeland (1708-1786) , Czech
Republic
292942 (- - - ) Poland
52023 (- - - ) Unknown Origin
243784 Ibatulla, 1860, Saya Kazanskiy uezd, Tatar ,
Russian Fed.
P278.2-y
(P278,
111 STRs or Big Y needed)
221114 Raspopin , Russian Federation
231126 Martin Wojcik, b. 1773, Poland
9925 Bruno Papallo, b.abt 1720, MARTONE , Italy
11721 Antonio Papallo, b. abt. 1872, MARTONE , Italy
317085 Andrew (Andras) Biro b. about 1860 Hungary
P278.2-y1 (more
STRs and/or P278 needed)
372707 Gawryszewski, Las , Poland
323637 Rado Junež d.1993 age 80, Slovenia
367657 b. abt. 1910, Debrecen, Hungary
161013 (- - - ) Unknown Origin
330920 John FIEDLER b. 1842 Bavaria Germany
234461 ст.Воровсколесская, Кубанский Козак , Russian Fed.
232447 (- - - ) Hungary
306789 Janos Kasper, b. 1818 Slovakia, d. 1877 Slovakia
155307 Lőcsei , Hungary
139888 Vyacheslav A. Bednyakov, b. 1859, d. 1906 ,
Russian Fed.
46335 Johann Strohmeier, b.1813, Pilsting , Germany
YP5281
NN
YP1702
B7738 Joseph Sawicki, b1740's Krzywcze, Borszczow,
Poland
YP371 (5 snp),
formed 2800-2300 yBP
YP371*
359328 (- - - ) Hungary
YP371*-A-x
(YP371 and/or Big Y needed)
272683 (- - - ) , Unknown Origin
317343 Jose Luis Fransua or Francois, b. abt 1850 ,
France
279653 Andrew Smith(Smit?)1864 Ostrobothnia 1909 ,
Finland
YP371*-x
(Big Y needed) [YP372-,YP971-]
52002 Paul Vojticek,b. 1881 and d. 1946 , Poland
207192 Stepan Senkevich, born c. 1850 , Ukraine
425156 Adopted - Ukrainian father , Ukraine
412231 Casimir Felckowski, ca 1720 - 1778 , Poland
YP371*-x1
(YP372, YP971 and Y11162 not tested, BigY needed)
251848 James Guthrie, b. 1775CT-d. 1850NY Scotland
YP971
N123777 John Kinnard, b 1734 Scotland, d 1800 S.C., U.S.,
Scotland
225697 Maciej Rapcewicz b. 1792 (Brańsk) , Poland
YP971-x (Big Y
needed)
355792 Krivosheev Mikhail, b. 1900 , Russian Federation
Y11162
331278 Robert Prater 1866-1926, Sumter O Prater b1897 ,
Unknown
B6861 Robert Prater 1866-1926, Eugene C Prater b1890,
Unknown
YP372 (4 snp),
formed 2300-1500 yBP
YP372*
B3415 August W. Tafel b.1837, Ulm; d. 1919, Chicago,
USA , Germany
E16481 ( - ) Macedonia
Y10810
- Russian
Federation
- Ryssian Federation
YP380 i YP379, YP374, Y6073, Y10810; formed 1500-1200 yBP (500-800 AD)
YP380*
E6498 Franz Brunner 1928-2014 Wolfsberg Carinthia ,
Austria
N116699 Andreas Staniek b.c. 1790 Poland
N49064 Christopher Galovič, b 1772, Matiašovce, Slovakia
57872 Johann Friedrich Rauch; 1807; Rudolstadt,
Germany
YP380*-x
(Big Y needed)
N37712 Stanley Simrajh, Maribor, Slovenia
260214 Karpovič , Belarus
YP380*-x1
(Big Y or Z280 SNP pack needed)
213870 Oskar Manty,1872-1949,Jalasjärvi,Finland
197855 Isak Esanpoika Laturi, 22.7.1788,Kauhajoki,Finland
214137 Debreczeny Mihály (ca.1600-1680) , Hungary
161093 Mihály Jász, 1672, Jászapáti , Hungary
YP380*-x2
(Big Y or Z280 SNP pack needed)
[another possibility: Z92>Z685>YP270>YP351]
249141 (- - - ) Poland
YP380-A
(BigY needed)
108620 Joannes Wodziak 1750-1797, Bialka Tatrzańska, Slovakia/Poland
99124 Stephanus Vodzak 1790-1850, Bialka Tatranska, Slovakia/Poland
YP380-z
(67 STRs needed)
N66118 Carlo Bogni, 1912-1999 Varese Lombardy , Italy
182802 Józef Guziewicz, b. 1844 and d. 1896 , Poland
189990 (- - - ) Poland
166081 Michael Odoliński, b.c.1739, Poland
210941 Gramatikovo, Bulgaria
YP4700, YP4701, YP4702,
YP4703, YP4704, YP4705;
formowanie: 800-1816 AD
169059 Maciej Pietrzak, 1766-1816,
Łukowica, Poland
177386 Andrzej Pietrzak, 1836-1900, Łukowica, Poland
YSEQ4524 Stanisław Pietrzak Jr
Łukowica/Jastrzębie, Poland
YSEQ4525 Paweł Pietrzak, Łukowica, Poland
YSEQ4526 Janusz Pietrzak, Łukowica, Poland
YSEQ4527 Mieczysław Pietrzak, Łukowica, Poland
|
|
Wszystkich członków rodu
YP343
zidentyfikowano dotąd 118
Według kraju pochodzenia przeważa Polska Południowa
i kraje na
południe od Karpat ku Italii.
Polska --- 32
Węgry ----14
Rosja ---- 13
Niemcy --- 7
Ukraina --- 7
Słowacja -- 6
Słowenia -- 4
Włochy ---- 4
Finlandia --- 4
Czechy----- 3
Austria ----- 2
Chorwacja - 2
Białoruś ---- 2
Szkocja ---- 2
Bułgaria ---- 1
Macedonia -1
Turcja ------1
Szwajcaria --1
Francja -----1
Anglia ------1
Dania -------1
Nieznane -- 10
..........
Badania genetyczne sugerują nam, że w
linii prostej mamy przodka Stefana Sienkiewicza,
z rodu R1a-YP371, który był bratankiem i siostrzeńcem Henryka
Sienkiewicza,
pisarza i noblisty.

::::::::::::::::::::::::::::::::::::::::::::::::::::::::::::::::::::::::::::::::::
A może i Ty w swoim Y-DNA masz snip
YP380 ?
Wykonaj niedrogie badanie (17,50 $ ) tego snipu w pracowni
YSEQ:
http://www.yseq.net/product_info.php?products_id=21486
Mogę pośredniczyć w załatwieniu prostych formalności i
przekazaniu próbki
do badania
:::::::::::::::::::::::::::::::::::::::::::::::::::::::::::::
Ustalenia z dnia 22.12..2015
Snipy, które w rodzie YP380 wyróżniają linię łukowickich Pietrzaków,
to
YP4700, YP4701, YP4702, YP4703, YP4704,
YP4705
Kolejne testowania genetyczne wykazały, że prawdopodobnie wszyscy Pietrzakowie
z Łukowicy i jej gminy
wyróżniają się snipem YP380. Więc może także tymi młodszymi snipami z serii
YP4700. Warto zbadać i przyłączyć się do naszego rodu!
|
* * * * *
Czytaj także o pochodzeniu innych grup ludności
w rejonie Dunajca: od Wisłoki do
Raby, od Tatr do Wisły
* * * * *
Można przypuszczać, że
archeologicznym dziełem naszych starokarpackich przodków z mutacją
YP343, żyjących
tu od około 3200 lat (oczywiście nie bez współudziału innych,
ojcowskiej i bratnich grup, np. Z280, CTS1211, CTS3402 i M458)
był obronny gród kultury wczesnego brązu
Otomani-Füzesabony na Górze Zyndrama
w Maszkowicach nad Dunajcem
i inne naddunajeckie punkty tej kultury.
Zob. na stronie
Stara karpacka gałąź rodu R1a
Kultura
Füzesabony-Otomani pojawiła się
około 2000 lat przed Chr. (wczesny brąz) w Kotlinie Karpackiej i nieco później na wzgórzu
Trzcinica koło Jasła, a zwłaszcza w grodzie w
Maszkowicach. Gród ten trwał wyjątkowo długo, bo przez blisko
2000 lat. Związane z nim były nieliczne i niewielkie osady otwarte, położone w pobliżu
Maszkowic i Łącka (więc zapewne w Łukowicy też). Grodzisko to aktualnie
(2011/2014) bada zespół archeologa dr Marcina Przybyły (Instytut
Archeologii UJ Kraków). Czytaj.:
http://www.archeo.uj.edu.pl/przybyla .
Nie dziwi więc,
że jedyny dość bliski (dotąd najbliższy) genetycznie mężczyzna
został zidentyfikowany we wschodnich Węgrzech, w miasteczku
Jaszapati, w powiecie Jaszbereny, w prowincji Jaszsag, o
nazwisku Michaly Jasz (czytaj: Jas).
.......................................................................................
..........................................................................................
UWAGA!
Genealogia genetyczna Y-DNA (i mitochondrialna mtDNA) liczy dopiero
około dziesięciu lat. Stale się rozwija, ubogaca, a zarazem zmienia. Kto
chce na bieżąco śledzić jej rozwój, proszę zaglądnąć na inną naszą stronę:
Skąd pochodzą Polacy, Słowianie,
Europejczycy i inne ludy?
|
Zachęcam także innych Pietrzaków i nie-Pietrzaków do
oddania swoich genów do badania,
aby wzbogacić wiedzę o ludności naszego
regionu i o wielkiej rodzinie słowiańskiej.
Udzielę także potrzebnych informacji praktycznych co do tych
badań.
Szczegóły, jak przystąpić do badania swojego Y i mtDNA
zob. na str.
Skąd Polacy. |
* * * * *
Tabela haplotypu STR rodu Macieja Pietrzaka
Poniższa tablica, prezentując
67-markerowy haplotyp
mutacji STR Macieja Pietrzaka, wskazuje:
w pierwszym wierszu - numer kolejny badanego markera (loci),
w drugim - oznaczenie segmentów badanych laboratoryjnie markerów DYS (czyli DNA Y-chromosome Segment),
w trzecim wierszu - liczbę alleli, czyli ilość powtórzeń krótkich tandemów
nukleotydów (STR - Short Tandem Repeat).
Cały zestaw alleli stanowi o haplotypie osoby. Allele są wykorzystywane do
porównywania haplotypów i rozpoznawania mutacji.
Dokładne miejsce naszego rodu YP340-YP380 na linii od Y-Adama
do Pietrzaków
i wśród innych rodów świata
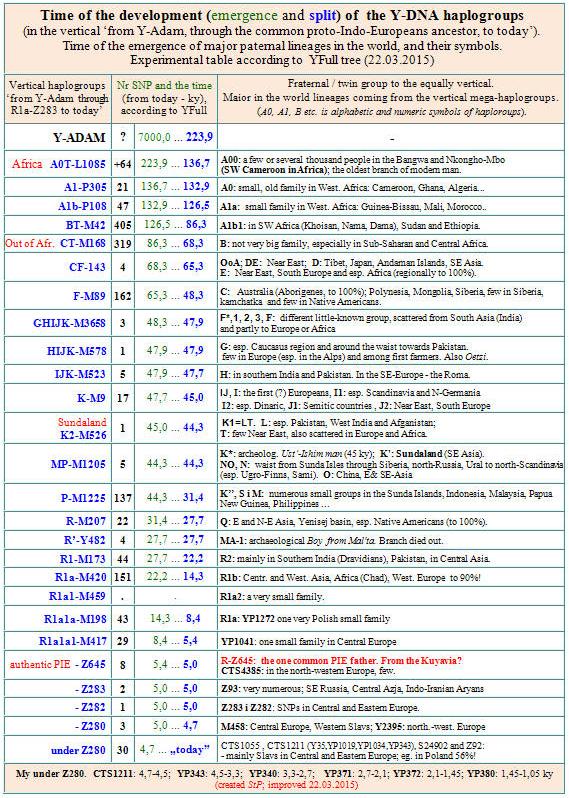
http://www.tropie.tarnow.opoka.org.pl/Genetyczne.rody.Y-DNA.w.swiecie.pdf
* * * * * * * * * * * * * * * * * * * * * * * * * * * *
* * * * * * * * * * * * * * * * * * * * * * * * * * * * * * * * *
Mitochondrialne pochodzenie
rodu Marii
Pietrzak od pierwszej matki - mitochondriaslnej "Ewy".
Droga naszych przodkiń od biblijnego Raju (w Afryce) do Łukowicy !
Drzewo genetyczne mitochondrialnego (matczynego) DNA,
począwszy od matki grupy L0 i L1 ("Ewa").
Jej czas życia, czyli TMRCA, obliczono na 177.000 lat.
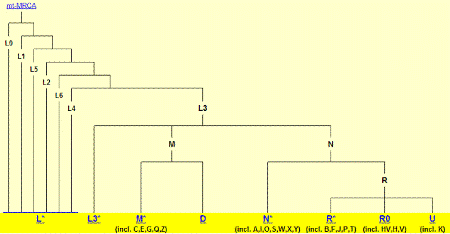
Haplogrupy L są rdzennie afrykańskie;
Haplogrupa L3 to głównie matka Azjatek
południowych i wschodnich i Amerykanek
Haplogrupa M to mtDNA Azjatek
Haplogrupa N to mtDNA Azjatek zachodnich i Europejek.
Na stronie źródłowej jest wejście do wszystkich podgrupL, L3, M, D, N, R, RO
i U,
według publikacji Behar et al. z kwietnia 2012 r.:
http://www.phylotree.org/tree/main.htm
Geny mitochondrialne (mtDNA) służą w organizmie oddychaniu i procesom
energetycznym.
Przekazywane są tylko przez matki i wyznaczają
matczyną genealogię każdego człowieka
- mężczyzny i kobiety - od pramatki,
mitochondrialnej Ewy.
Poniższa haplogrupa mtDNA dotyczy tylko mojej linii matczynej,
prababci Katarzyny Iwan Szlagowej z Łukowicy, wszystkich jej potomków żeńskich, a z
męskich - tylko ich synów (czyli i mnie).
Synowie otrzymują mtDNA od matek,
ale nie mogą go przekazywać swoim potomkom.
Zob. więcej na stronie
Szlagowie.
Od Katarzyny Iwan geny mitochondrialne odziedziczyła Franciszka
Szlaga-Janikowa,
a od niej Maria Janik-Pietrzakowa, moja matka.
Od
niej zaś - wszystkie jej dzieci, łącznie ze mną.
Drzewo
mitochondrialnej haplogrupy mtH i w nim gałąź H14a
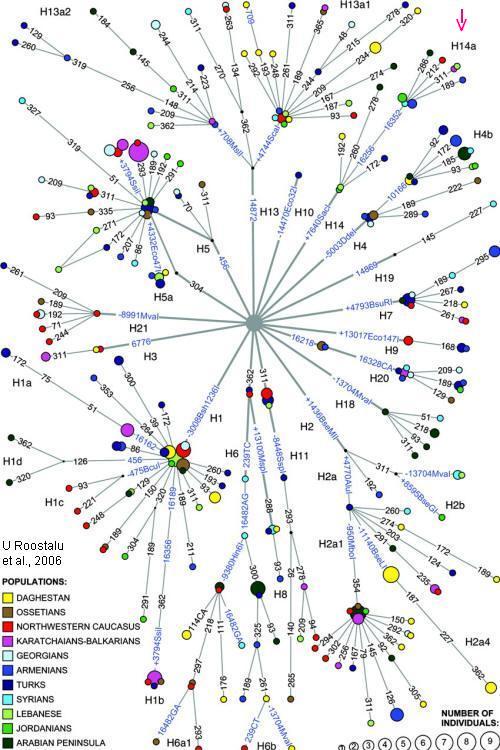
|
Zob.
kolejne mutacje i haplogrupy : mtEwa, L3, -N-R-VH--H--H14--H14a. |
Moja HAPLOGRUPA
matczyna,
mtDNA:
H-14a
Matczyna haplogrupa mtDNA,
odziedziczona
przeze mnie od Katarzyny Iwan Szlaga z Łukowicy,
a przez nią od jej matki Marii
Rzepka/Dudka/Iwan i wszystkich
poprzednich pra-matek, to haplogrupa H14a.
Powstała ona w Jordanii, w trakcie migracji grup ludzkich od mitochondrialnej Ewy z Środkowo-Wschodniej Afryki,
przez Arabię, Bliski Wschód, Jordanię, Liban i Armenię ku Kaukazowi.
W Jordanii w wielkiej haplogrupie H
( litera H - od imienia Helena) powstała bardzo mała haplogrupa H14a.
W rejonie Kaukazu haplogrupa H, a z nią i daleka jej
córka H14a, brała udział w kształtowaniu ludzkiej rasy
kaukaskiej, europidalnej, czyli białej. Po przejściu
Kaukazu dalsza migracja ku Małopolsce szła zapewne przez Ruś południową
i Węgry lub Podkarpacie.
W Europie,
Polsce i Małopolsce
mitochondrialna haplogrupa H to około 40% matek i ich
synów, a naszej H14a - to
zaledwie ułamek procenta
(aktualnie zidentyfikowano tylko kilka osób H14a na
około 10000 przetestowanych w
świecie).
Spośród świętych
Kościoła haplogrupą H odznaczał się np. św. Łukasz Ewangelista (co właśnie udało się zbadać, zob. C.Vermesi et al., 2001), a
spośród ludzi mniej świętych - austriacki cesarz Józef II i cesarzowa Maria Teresa.
|
UWAGA!. Naszą haplogrupę mitochondrialną
H i jej podgrupy, w tym także H14a, naukowo opracował zespół prof.
U.Roostalu i jego zespołu z Estonii i Rosji, w pracy:
Pochodzenie i ekspansja mitochondrialnej haplogrupy H, 2007
(w j.angielskim).
|
Osiem
tysięcy praojców
i tyleż pramatek mam.
Zapadły się ich mogiły
i nie ma ich kości ...
Ale znajduję coś w sobie
- ich własne de-en-a.
Poznam
je, przy nim uklęknę
w synowskiej miłości!
....................................................
Moje mutacje: od
Y-Adama: L74
od Macieja Pietrzaka: R-YP4700
od Franciszki Szlaga: mt-H14a |
Informacja i prośba do Pietrzaków z Łukowicy
(lub Stańków z Cieszyńskiego)
w sprawie genetycznego pokrewieństwa
Wszyscy Pietrzakowie z parafii Łukowica mogą być ze sobą spokrewnieni, ale
dotąd jesteśmy pewni na podstawie metryk tylko co do rodu Macieja w linii jego
wnuka Andrzeja Pietrzaka z Przedewsia (Łukowica 31). Dla innych brak
wystarczająco wczesnych zapisów w księgach niż z lat około 1750. Ci więc Pietrzakowie, których
interesuje pokrewieństwo z Pietrzakami z Przedewsia mogą wykonać odpowiednie
badania genetyczne. Proszę więc, aby męscy przedstawiciele pozostałych
łukowickich rodów Pietrzaków wykonali sobie takie badania genetyczne.
Badanie genetyczne dla tych łukowickich
Pietrzaków ,
którzy chcą wiedzieć czy należą lub
nie należą do naszego rodu
Pietrzakowie z
parafii Łukowica oraz z okolicznych wsi mogą zbadać swoje męskie pokrewieństwo z
Pietrzakami z Łukowicy - Przedewsia.
W firmie Y-SEQ Thomas Krahn http://www.yseq.net/. Można zamówić zbadanie
tylko jednej mutacji SNP, która wyróżnia Pietrzaków z Przedewsia,
mianowicie YP380, za 17,50$. Mutacja
YP380 wskaże przynależność do
tych Pietrzaków,
a jej brak zaprzeczy tej przynależności - nic więcej, Jest
to pokrewieństwo z Pietrzakami dawne jak historia
Polski, a nawet do około 1200 lat od wspólnego przodka.
Ale bardziej dokładny zakres:
stopień i czas pokrewieństwa wskaże badanie sześciu mutacji
(SNP) w takim mini-pakiecie: YP4700, YP4701, YP4702,
YP4703, YP4704 i YP4705.
Zamów na stronie:
http://www.yseq.net/product_info.php?manufacturers_id=33&products_id=23348
lub
http://www.yseq.net/index.php
Kliknij zamówienie: Buy Now
(Uwaga; powtórne kliknięcie podwoi zamówienie 1>2;
wtedy trzeba je cofnąć!). Wymień wszystkie 6 mutacji z grupy
YP.
Ustal sposób zapłaty: kartą płatniczą lub inaczej, np.
PayPal (mogę w tym pośredniczyć).
Firma przysyła tzw. kit, czyli instrumentację do zrobienia
wymazu z jamy ustnej, hermetycznego zapakowania i wysyłki
zwykłą pocztą pod wskazany adres:
YSEQ, A. Krahn
Eiswaldtstr. 31
12249 Berlin
GERMANY
http://www.yseq.net/product_info.php?products_id=21486

Mogę pośredniczyć w zamówieniu tego badania.
xStanisław Pietrzak
....................................................................................
..................................................................................
Dla innych, nie Pietrzaków, taka jest najlepsza możliwość
badań genetycznych.
Po kliknięciu
https://www.familytreedna.com/y-dna-compare.aspx trzeba zamówić badanie
wstępne: testowanie 37 markerów STR Y-DNA za 169 $. (albo od razu 67
markerów STR). Tam wypełnia się
formularz zgłoszeniowy i ustala sposób płatności; firma przysyła kit czyli
jakby szczoteczkę i pojemniczki na wymaz z jamy ustnej. Po odesłaniu i kilku tygodniach
firma podaje informacje o wynikach: podaje zestawienie mutacji i z grubsza
przynależność do konkretnej grupy.
Ten wynik można zgłosić do
"Polish Project FTDNA", którego administratorem jest Amerykanin polskiego
pochodzenia, Lawrence Mayka
https://www.familytreedna.com/groups/polish/about/background (tam jego
E-mail; można pisać po polsku, choć odpowiada po angielsku), podając numer #
kit. On po dokładniejszej ocenie i ustaleniu gałęzi genetycznej wprowadzi
wynik w tabelę tegoż Polish Project, a jeżeli okaże sie haplogrupa R1a, to
przekaże ją na specjalny portal dla tej grupy "R1a1a and Subclades", której
administratorami są także dr Łukasz Łapiński i dr Michał Milewski;
https://www.familytreedna.com/groups/r-1a/about/background
Oni mogą
wskazać drugi etap badań na dwa sposoby: albo pełne badanie w programie BigY (cena w okresie
promocji około 400-450$), albo uproszczone i tańsze w wybranym pakiecie (w
cenie około 100$). To umożliwi umieszczenie gałęzi na światowym drzewie
genealogicznym rodu R1a. YFull tree:
https://www.yfull.com/tree/R1a/
|



























